使用Aspera加速SRR下载
IBM Aspera 是为远距离大文件传输量身定制的传输工具。它基于 UDP 改造,在提高下载速度的同时保证数据质量。
IBM Aspera 是为远距离大文件传输量身定制的传输工具。它基于 UDP 改造,在提高下载速度的同时保证数据质量。
IBM Aspera 由 Server 和 Client 组成,Client 即 IBM Aspera Connect,是供所有人免费使用的。
生物信息学领域,原始测序数据的大小可达数十甚至数百 GB,且往往要跨大洲传输数据,面临高延迟(> 100ms)和高丢包率(> 5%)的挑战。再用传统的 TCP 可就力不从心了。IBM Aspera 则采用基于 UDP 的 FASP(Fast and Secure Protocol),利用 UDP 传输数据,而在应用层,通过多线程重传、链路加密与自适应带宽调节,在实现极高传输速度的同时,达到与 TCP 无异的高可靠性。
安装
在 Conda 的 HCC channel 里就可以安装 IBM Aspera Connect:
conda install -c hcc aspera-cli
也可以使用 apt/yum 安装,软件包的名字是一样的。
在这个网页可找到 IBM Aspera Connect 的下载链接。
使用
1
2
3
4
5
$ ascp -h
Usage: ascp [OPTION] SRC... DEST
SRC to DEST, or multiple SRC to DEST dir
SRC, DEST format: [[user@]host:]PATH
......
最常用的 OPTION 包括
| 参数/flag | 用途 |
|---|---|
-T | 禁用加密。非敏感数据请使用该 flag,可改善下载速度 |
-v | 启用详细模式 |
-l/-m | 最小/最大传输带宽(bps) |
-i | 私钥(id_rsa)位置。如果使用 SSH 免密,需使用私钥文件 |
-k | 断点续传等级。使用 1 即可 |
--mode | 模式(下载 recv/上传 send) |
--user/--host | 用户名/主机名 |
ENA(European Nucleotide Archive)对 Aspera 的支持性比 NCBI SRA 的好一点,所以先用 ENA 做示范,介绍如何批量下载 SRR 数据。
获取 URL
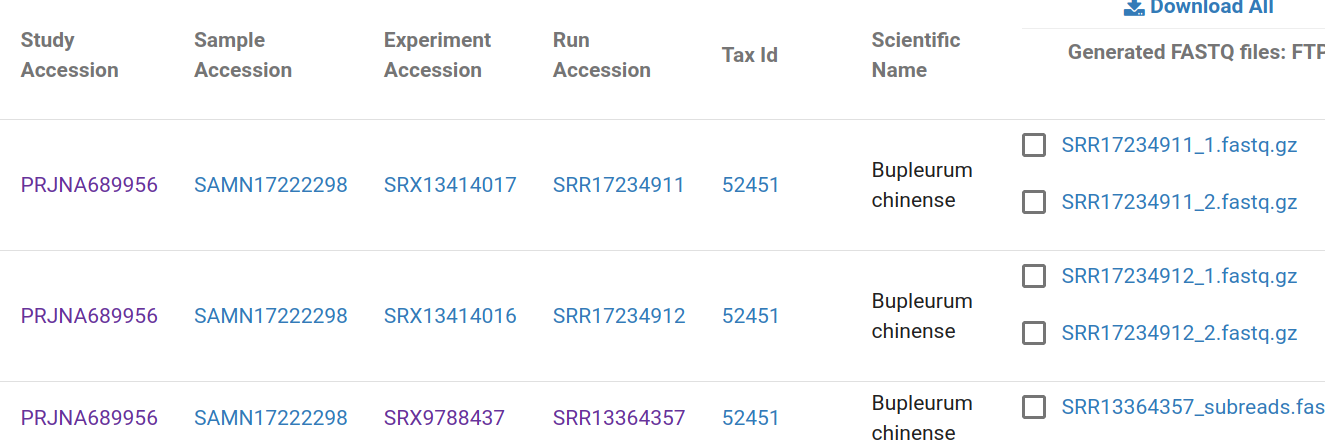
建议查 PRJNA,而不是 SRR,虽然二者都会在论文里直接标注。不太建议单查 SRR,到时候要一个个找,找起来会很痛苦。PRJNA 底下就有一整套的 SRR 条目。
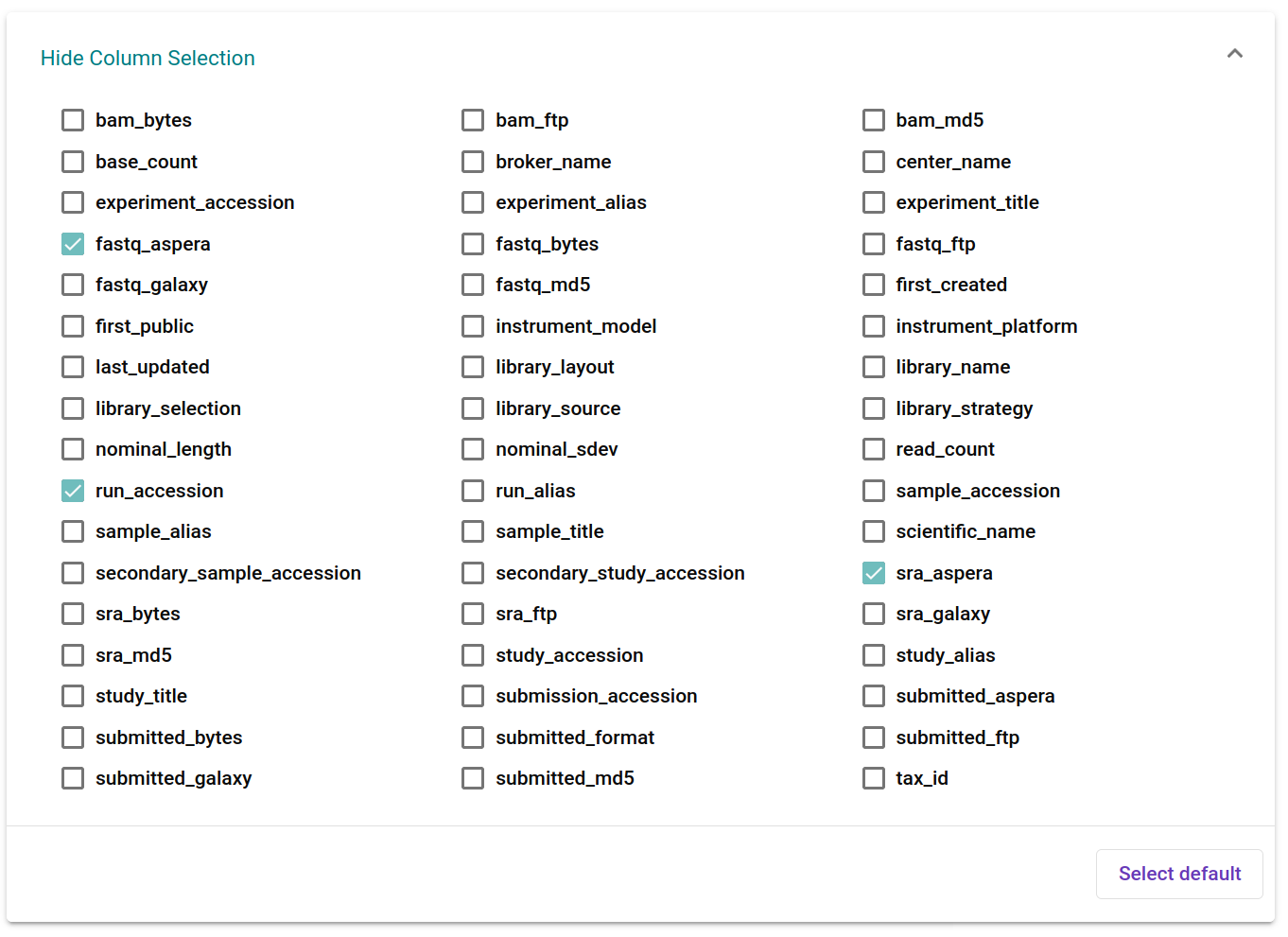
然后,点开上面的“Show Column Selection”,只给“run accession”和带“aspera”后缀的打勾。
我比较喜欢直接下 fastq,省去从 SRA 转换成 fastq 的麻烦。所以只点“run accession”和“fastq_aspera”。
在 Download report 那里,点“tsv”。
就能得到一系列的 url:
1
2
3
4
5
run_accession fastq_aspera
SRR17234911 fasp.sra.ebi.ac.uk:/vol1/fastq/SRR172/011/SRR17234911/SRR17234911_1.fastq.gz;fasp.sra.ebi.ac.uk:/vol1/fastq/SRR172/011/SRR17234911/SRR17234911_2.fastq.gz
SRR17234912 fasp.sra.ebi.ac.uk:/vol1/fastq/SRR172/012/SRR17234912/SRR17234912_1.fastq.gz;fasp.sra.ebi.ac.uk:/vol1/fastq/SRR172/012/SRR17234912/SRR17234912_2.fastq.gz
SRR13364356 fasp.sra.ebi.ac.uk:/vol1/fastq/SRR133/056/SRR13364356/SRR13364356_1.fastq.gz;fasp.sra.ebi.ac.uk:/vol1/fastq/SRR133/056/SRR13364356/SRR13364356_2.fastq.gz
......
整理 URL
一个 SRR 条目往往有多个 URL。为此需要展开它们,然后将 URL 之外的东西删干净。——又该正则表达式出场了。
1
2
3
4
5
awk -F'\t' 'NR>1 {split($2, a, ";"); for(i in a) {
sub(/.*fasp\.sra\.ebi\.ac\.uk:/, "", a[i]);
print a[i];
}}' ena_aspera.tsv > url_list.txt
其中
| 部分 | 含义 |
|---|---|
awk | 是一个非常强大的文本处理工具(处理行、列、模式匹配) |
-F'\t' | 指定“列分隔符”为制表符(\t = Tab),因为这是一份 TSV 文件 |
'NR>1 { ... }' | NR 是当前行号(Number of Record)。NR>1 意思是跳过第一行 |
$2 | 表示第二列的内容(在这里是 fastq_aspera 这一列) |
split($2, a, ";") | 把第二列的内容(用分号分隔的多个 URL)拆分成数组 a |
for(i in a) | 对于数组 a 里的任何元素 |
sub(/.*fasp\.sra\.ebi\.ac\.uk:/, "", a[i]) | 删除fasp.sra.ebi.ac.uk:,并捕获该结果 |
print a[i] | 打印这个结果 |
> url_list.txt | 输出保存到 url_list.txt 文件里 |
批量下载
使用命令
1
$ ascp -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh -l 300M -v -k1 -P33001 --mode recv --user era-fasp --host fasp.sra.ebi.ac.uk --file-list url_list.txt .
需要注意:
-i是私钥文件的位置。使用 Conda 安装的,密钥在 Aspera Connect 所在 Conda 环境的etc文件夹下,名字同样叫asperaweb_id_dsa.openssh。批量下载时必须注明
--host,而--user--host已经在命令中声明的情况下,url_list.txt不应该包含用户与主机号,否则会出现No such file or directory错误。单文件下载则可以随意一些,不使用
--user--host,而是user@host:/path。
如果就是不想用 Aspera…?
NCBI SRA 提供了下载 SRR 的官方程序:SRA Toolkit。它可以用 Conda 安装:
conda install -c bioconda sra-tools
使用 prefetch 命令下载:
prefetch SRR17234911
不过,
prefetch默认只下载小于 20GB 的 SRR,除非手动输入-X参数。但即使是提高限制,下载过程也非常痛苦。我并不喜欢这种方法。
批量下载的话,可以将所有 SRR 写在一个 txt 里(SRR.txt),然后执行
prefetch --option-file SRR.txt
不想用 NCBI 公服,可使用 srapath 获得下载链接:
1
2
$ srapath SRR17234911
https://sra-pub-run-odp.s3.amazonaws.com/sra/SRR17234911/SRR17234911
可以看到,这个数据被托管在 Amazon Web Services(AWS)上。不用说,下载质量肯定比 NCBI 公服好很多。
NCBI 这几年与 Amazon Web Services(AWS)、Google Cloud Platform(GCP)积极展开合作,特别是将自己的 SRA 数据,以及经授权可公开访问的数据交给他们托管。这不仅方便人们下载,还可以利用他们的云上资源(AWS 的 Athena,GCP 的 BigQuery)直接进行检索与分析。
然后再 wget。
1
$ wget https://sra-pub-run-odp.s3.amazonaws.com/sra/SRR17234911/SRR17234911
也不是不行。
有时甚至效果更好。
将这些 SRR 转换为 fastq,需要使用 fasterq-dump。fasterq-dump 的好处是能利用多线程,加快解码速度(默认 6 线程,可使用 -e 参数重新设置)。想看进度条,请加 -p flag。
需要注意,对于双端测序数据,还要加一个 flag(取其中一个):
-s:双端测序结果分别放在两个文件里。-S:同上,但 unpaired 的部分直接丢弃。-3:unpaired 的部分放在第三个文件里。
如果想直接输出压缩档(gzip/bzip2),请用 fastq-dump。但这样就享受不到多线程的好处了。