使用WGDI重建祖先染色体核型及其演化过程
像解魔方一样有趣好玩,又令人恼火。
感谢川大孙朋川提供的WGDI(Whole-Genome Duplication Integrated)研究工具,以及使用该工具进行核型演化研究的帮助文档。原始文档可在此处获取。
什么是核型演化(Karyotype Evolution)?
核型(Karyotype)包含一个物种的染色体数目,以及每条染色体的特征。核型演化反映了从祖先基因组到现代基因组的染色体变化,这些信息对重建祖先核型、追溯现代物种如何逐步演化至关重要。
祖先染色体核型(Ancestor Chromosome Karyotype,ACK)的演化——尤其是涉及到染色体数量的变化——通常会导致物种形成,并伴随快速的生殖隔离。然而,在现有物种漫长的核型演化史中,祖先染色体的融合或断裂会在后代谱系中逐渐且随机地发生,这意味着每条完整的祖先染色体1可能在不同高度分化的谱系中得以保留。此外,相较于经历更多全基因组复制(whole-genome duplication,WGD)的远缘物种,基因组变化较少且更接近共同祖先的谱系可能保留更多的原染色体片段。
对于某个原染色体与另一个融合形成新染色体,或断裂成两个新染色体的行为,我们常常利用共线性区块(syntenic blocks)作为基本规则之一进行识别。不过,另一个经常被人忽视的基本规则是:所有原染色体或其共线性区块需与各后代物种的染色体完全对应。
识别 ACK 的基本规则与方法
以往进行的 ACK 重建,常采用基于共线性关系定义的共同祖先区域(contiguous ancestral regions,CARs)。然而,由于染色体结构反常,以及零散狭窄的共线性区段,未定义区域会形成间隙,导致结果高度依赖于输入文件与参数设置。大量的间隙区域还会阻碍对染色体变化的进一步探索。与传统方法不同,我们通过寻找高度分化谱系间染色体融合过程中共享的完整染色体或类染色体共线性区块,来鉴定ACK。
在染色体融合过程中,受端粒保护的类染色体同线性区块得以保留,使得融合后的染色体携带端粒结构。但需注意,本方法并未排除染色体分裂现象——即一个原始染色体可能分裂为两个染色体。在此情形下,我们可在其他谱系中识别该原染色体,其可能完整保留或与其他原始染色体发生融合。
我们归纳了三种基本染色体融合类型中的核型变化规律,并阐述了用于重建被子植物 ACK 的鉴定流程。
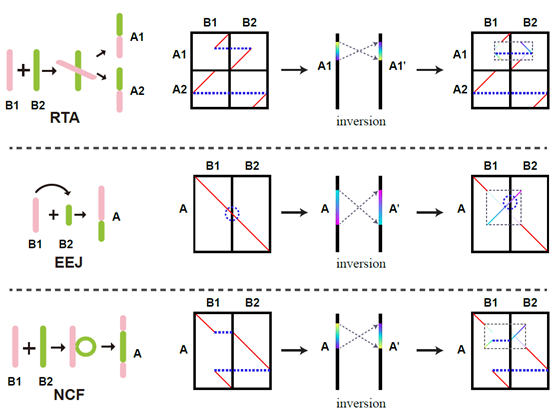
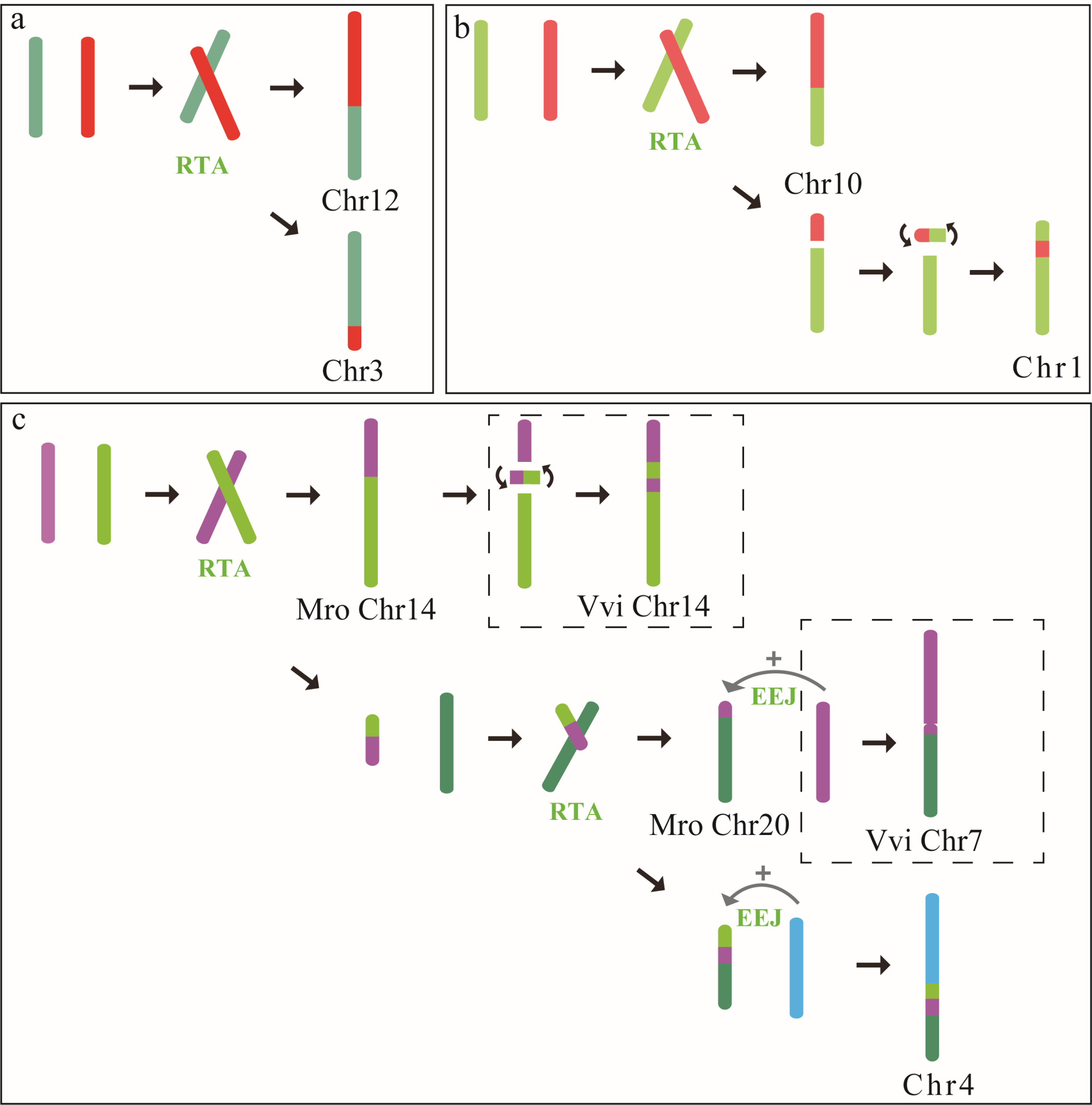
三种不同的染色体融合模式
基本的染色体融合模式(图1),包括相互易位染色体臂(reciprocally translocated chromosome arms,RTA)、末端-末端连接(end-end joining,EEJ),以及嵌套式染色体融合(nested chromosome fusion,NCF)。染色体易位包括相互易位和罗伯逊易位,分别对应 RTA 和 EEJ。NCF 模式也已被广泛认知并应用2。
图1 染色体融合的三种基本模式
尽管染色体结构变异在基因组中频繁发生,但原染色体融合断点恰好与后续进化阶段中其他结构变异断点重叠的概率似乎非常低。因此,这类融合断点可准确用于推断两个谱系是否拥有共同祖先。类似地,虽然倒位常在染色体进化过程中出现,但其几乎不可能破坏原染色体融合断点 。故倒位对核型重建的影响甚微。
染色体分裂通常指一个原染色体断裂成两个或多个独立染色体。上文定义的 RTA 属于特殊染色体分裂。其余染色体分裂未纳入考量基于两点原因:其一,全基因组加倍在植物中普遍存在,且染色体融合在全基因组加倍后更易发生;其二,若原染色体分裂发生于某一谱系(例如断裂成多条染色体),该原染色体的识别可能被其他谱系替代——在那些谱系中它可能保持完整或与其他原染色体以同线性区块形式融合。此外,两个分裂后的染色体同样对应同线性区块,这些区块可在原染色体识别的第二循环中被识别并删除。
构建 ACK 及核型演化历史的基本步骤
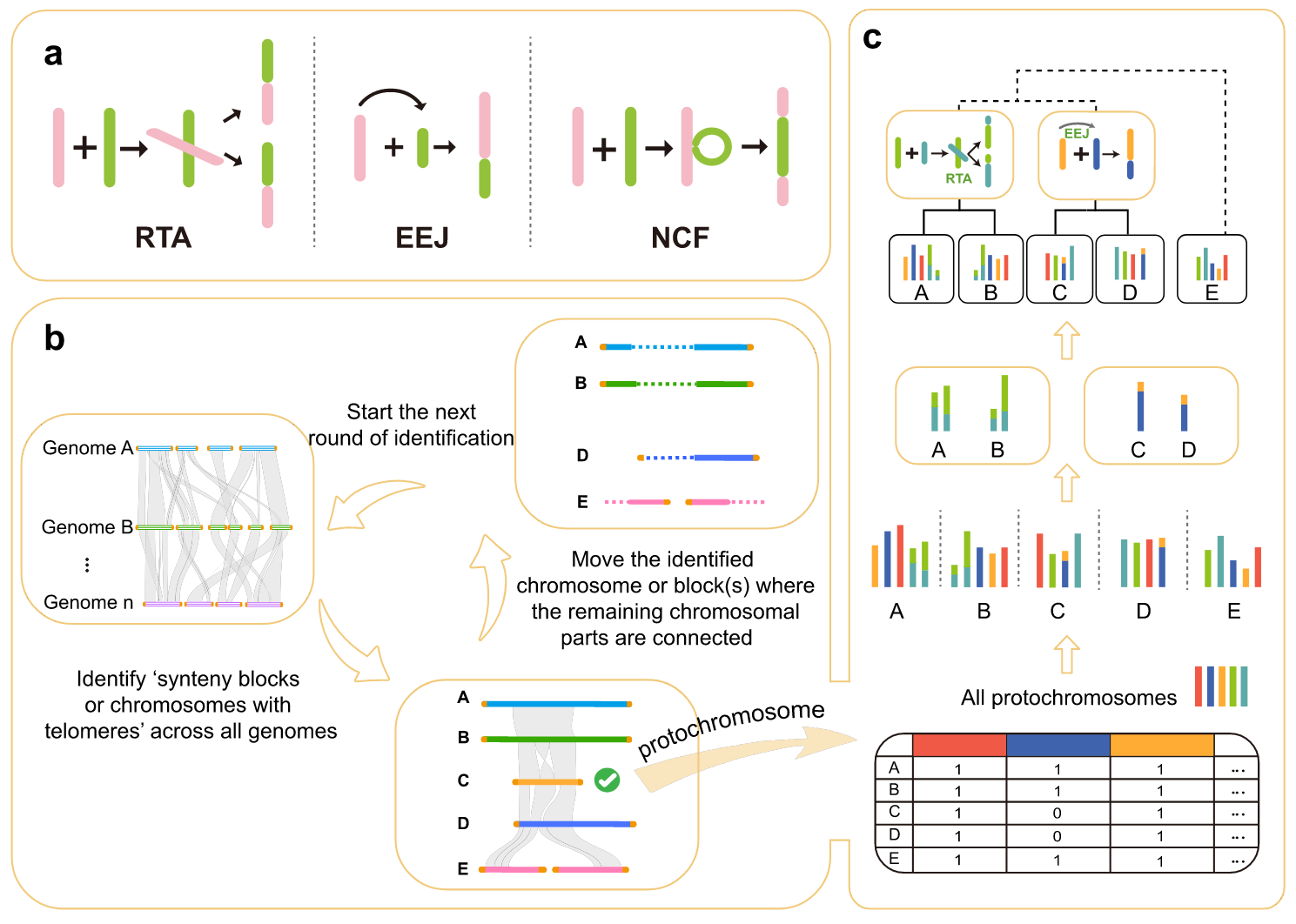
构建 ACK 的过程可分为四步(图2,a 和 b):
- 在所有采样基因组中识别类染色体”共线性区块”及小型共线区块。
- 探查现存基因组中与端粒共享的类染色体”共线性区块”及完整染色体,并从中提取最完整的单元(例如某条染色体)作为原始染色体。
- 在所有现存基因组中删除该共享区块及其同源小型区块,并将剩余部分连接为”完整染色体”。
- 启动新一轮”共线性探查”,直至每个现存基因组无剩余基因组区块为止,从而提取所有原始染色体。
简单说就是,从没有发生染色体变异的染色体出发,一步步地补全基因和染色体,组成祖先核型;再用这个祖先核型向其他物种比对。被选择的初始染色体全段最好与其他的染色体都有良好的共线性关系。
图2 根据三种基本的染色体融合类型(a),基于高度分化谱系间共享的染色体或类染色体同线性块来初步识别原染色体(b),并确定共同祖先的系统发育关系和核型进化(c)。
ACK 的所有原染色体均与每个现存基因组进行比较,并确定了这些原染色体组成的核型特征。根据 ACK 原染色体与现存物种染色体之间的融合和断裂位置,可推断核型变化及系统发育关系(图2c)。
核型重建演示——以 AEK 为例
AEK3 的染色体数目和基因组成几乎是公认的。我们以自己的方法为例,展示 AEK 的详细重建过程。
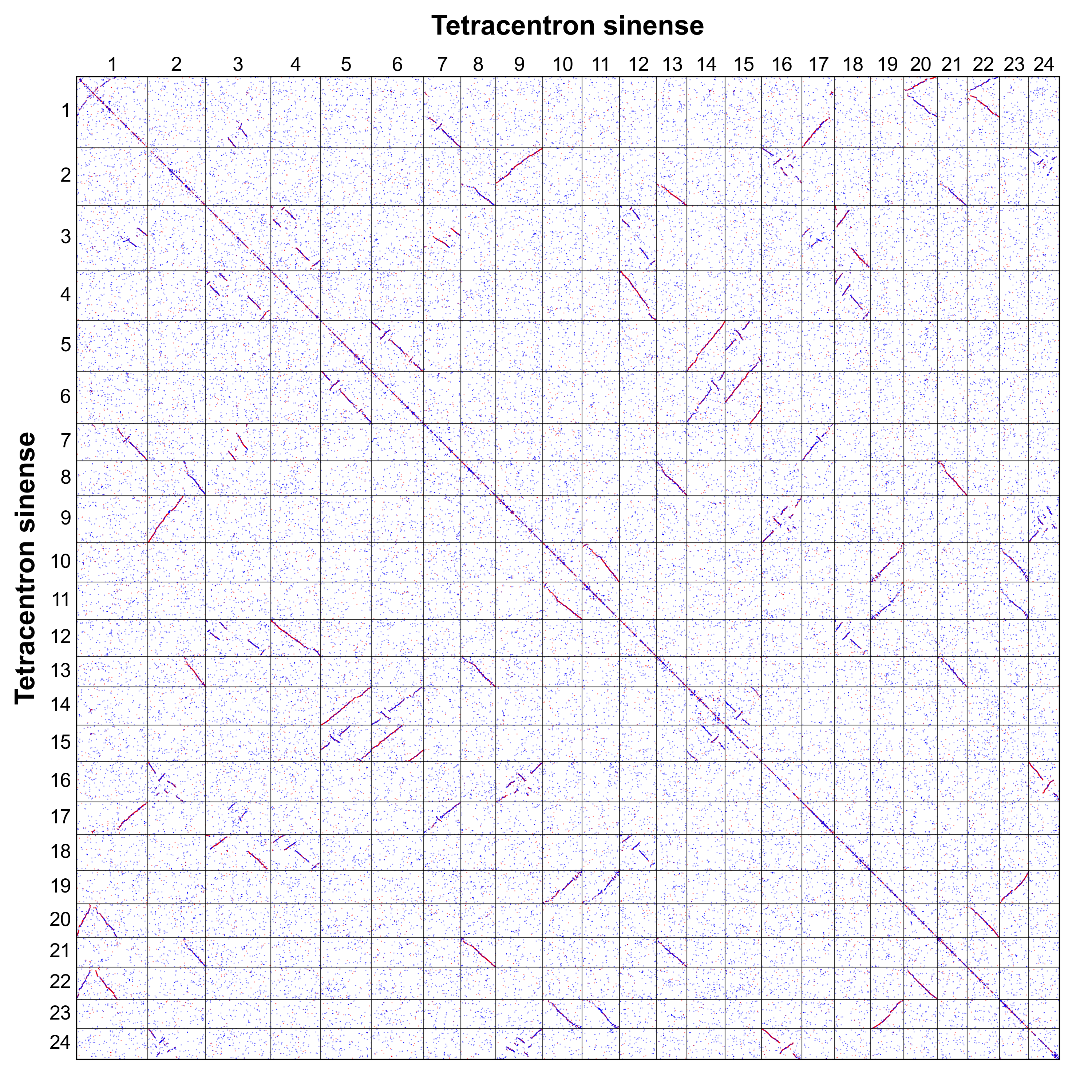
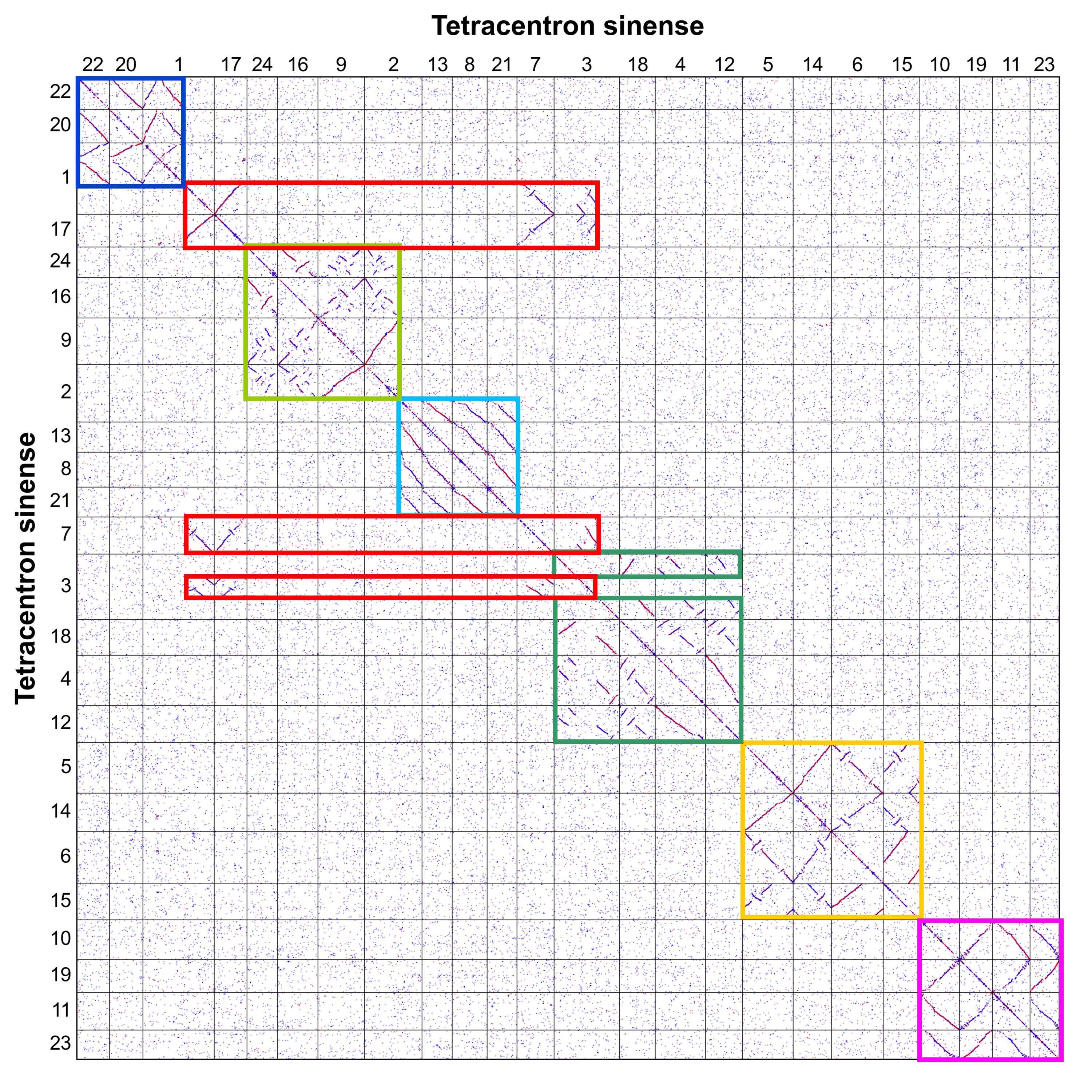
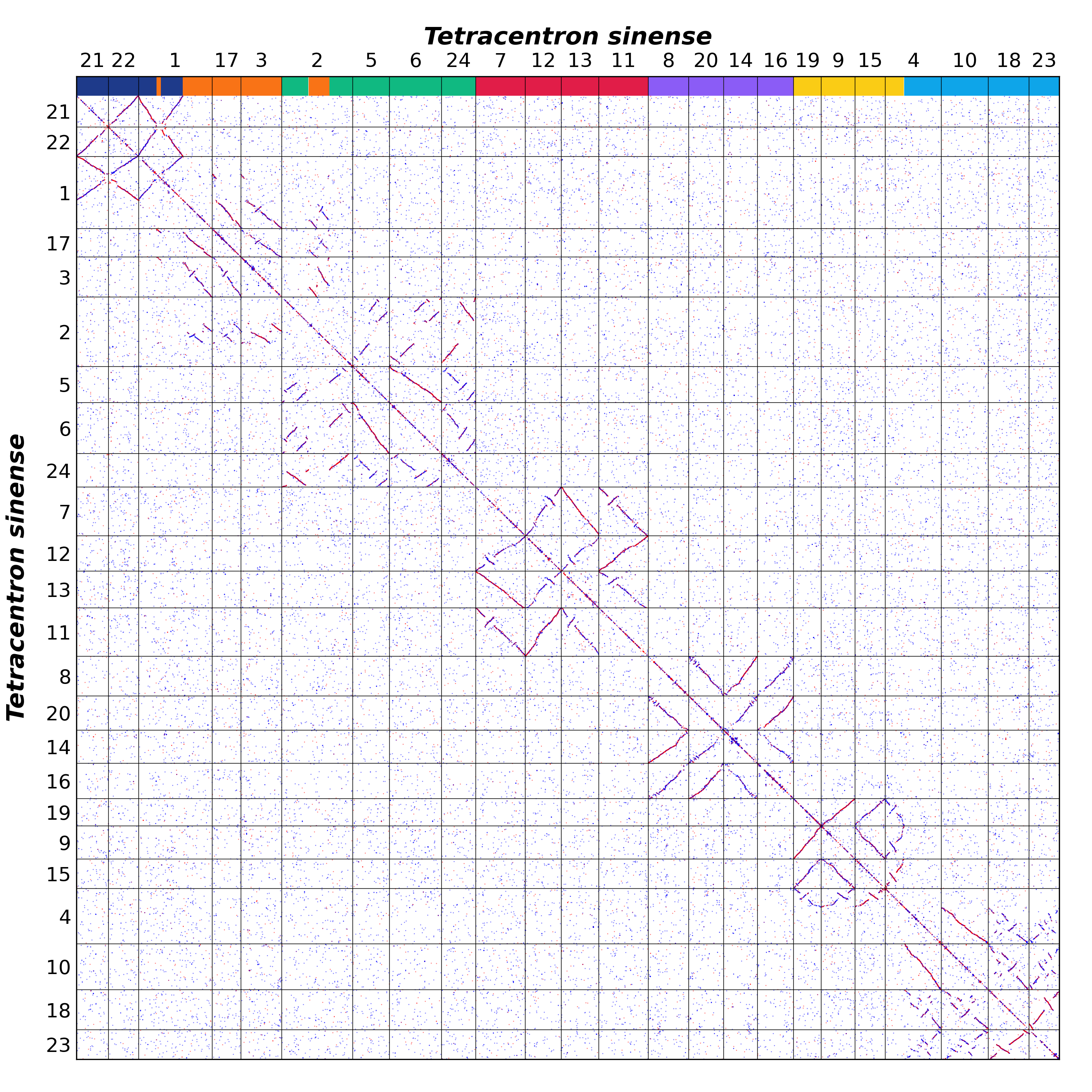
我们利用水青树(Tetracentron sinense)4重建 AEK。首先使用 WGDI 工具的 -d 参数绘制水青树内部的共线性 d-d plot。多倍化分析表明水青树经历了两轮全基因组复制事件,这意味着该物种保有 AEK 的四份拷贝(图3)。
图3 水青树基因组自共线性点图
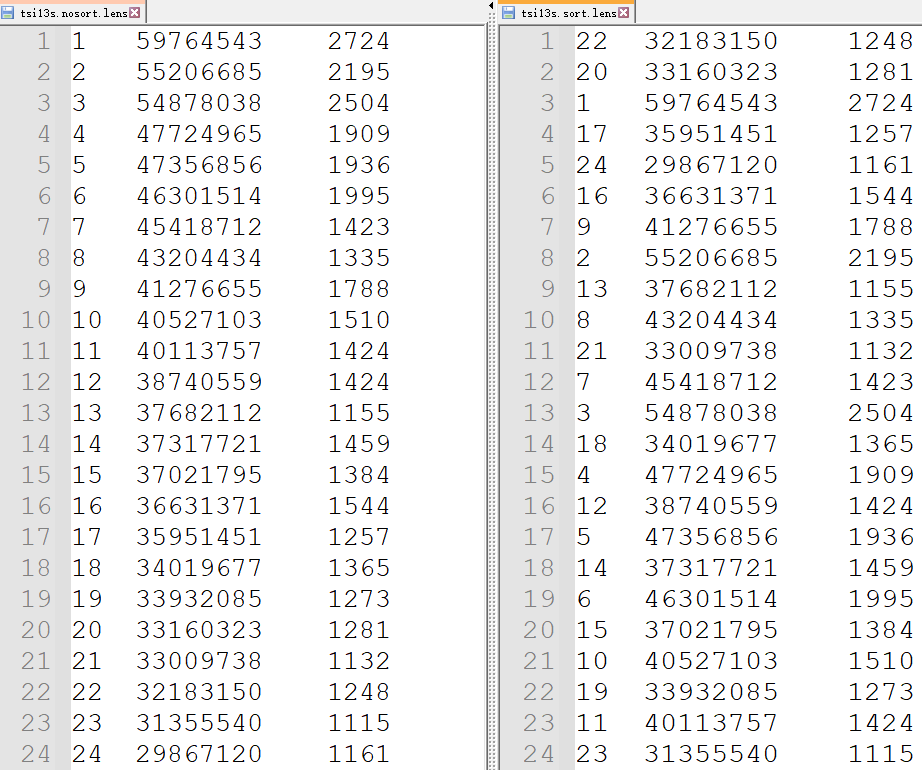
之后,我们在 .len 文件中反复调整染色体的顺序,让这些共线性连线各自聚到一起(图4和图5)。
图4 调整前(左)/后(右)的 .len 文件
图5 重新绘制的 d-d plot,可清楚看出其 1:4 的共线性深度。疑似来自不同原染色体的共线性区段已在图中用不同颜色标出
我们分别从不同簇群中以完整染色体作为原染色体提取单倍型。例如蓝色簇中的 22 号和 20 号染色体、红色簇中的 17 号和 7 号染色体、草绿色簇中的 24 号、16 号和 9 号染色体、天蓝色簇中的 13 号、8 号和 21 号染色体、深绿色簇中的 18 号、4 号和12 号染色体、橙黄色簇中的 5 号、14 号、6 号和 15 号染色体,以及紫色簇中的 10号、19号、11号和23号染色体。我们从每个簇群中选取一个原初染色体,并用不同颜色和组别进行标记。结果文件如下所示(第一行不在原文件里,只是为了放止加粗而已)。
| 22 | 1 | 1248 | RoyalBlue | 1 |
| 17 | 1 | 1257 | red | 1 |
| 24 | 1 | 1161 | #99CC00 | 1 |
| 13 | 1 | 1155 | deepskyblue | 1 |
| 18 | 1 | 1165 | #339966 | 1 |
| 5 | 1 | 1936 | #FFCC00 | 1 |
| 10 | 1 | 1510 | fuchsia | 1 |
再利用 -ak 参数提取该基因组的 AEK 单倍型。AEK 文件如下所示(第一行同样不在原文件里)。
| 1 | 1 | 1248 | RoyalBlue | 1 |
| 2 | 1 | 1257 | red | 1 |
| 3 | 1 | 1161 | #99CC00 | 1 |
| 4 | 1 | 1155 | deepskyblue | 1 |
| 5 | 1 | 1165 | #339966 | 1 |
| 6 | 1 | 1936 | #FFCC00 | 1 |
| 7 | 1 | 1510 | fuchsia | 1 |
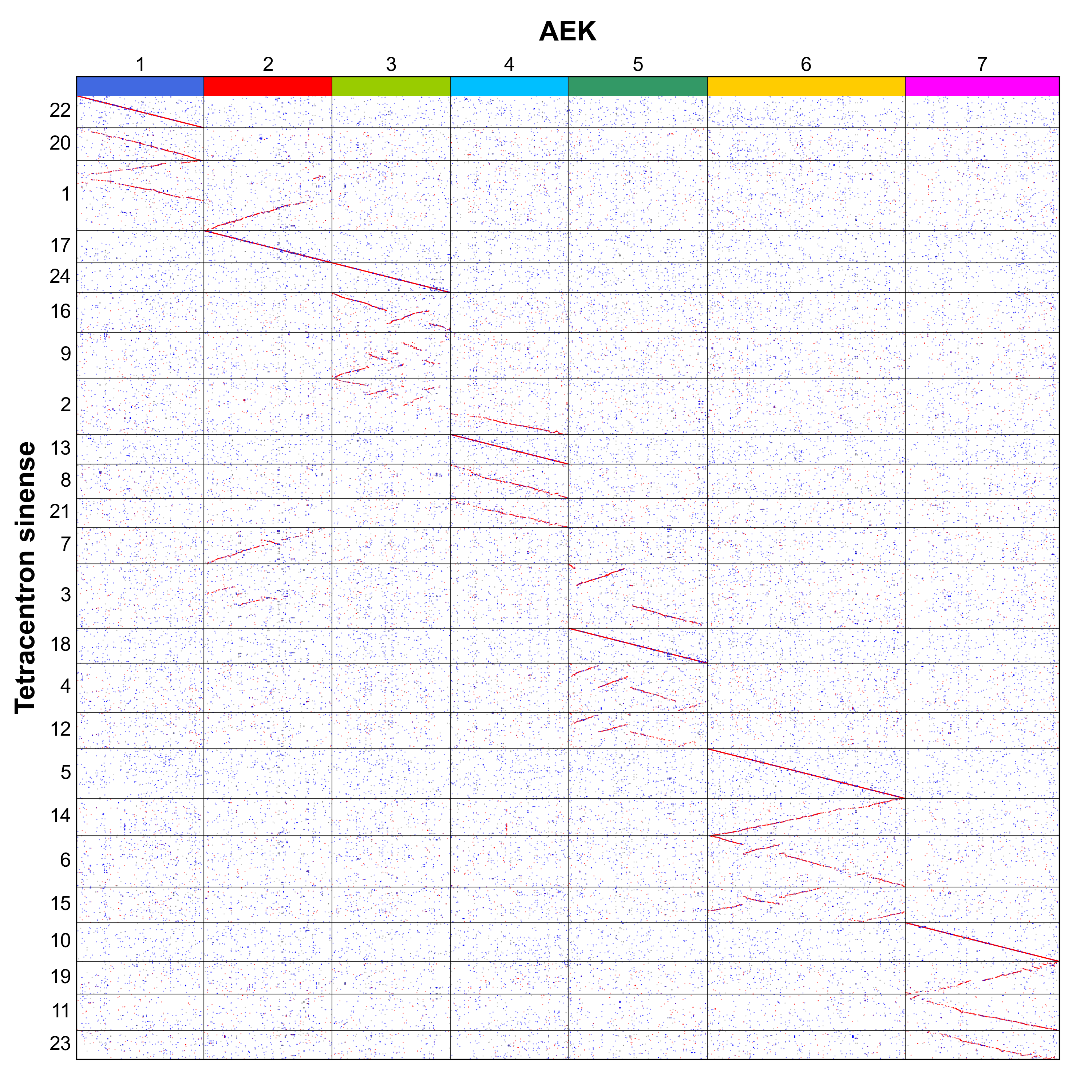
水青树的大部分基因应源自 AEK。为防止忽略其他原染色体,我们进一步将 AEK 映射至水青树。首先采用 WGDI 工具的 -d 参数绘制水青树与 AEK 的同源 d-d plot(图6)。
图6 水青树与 AEK 的共线性关系点图
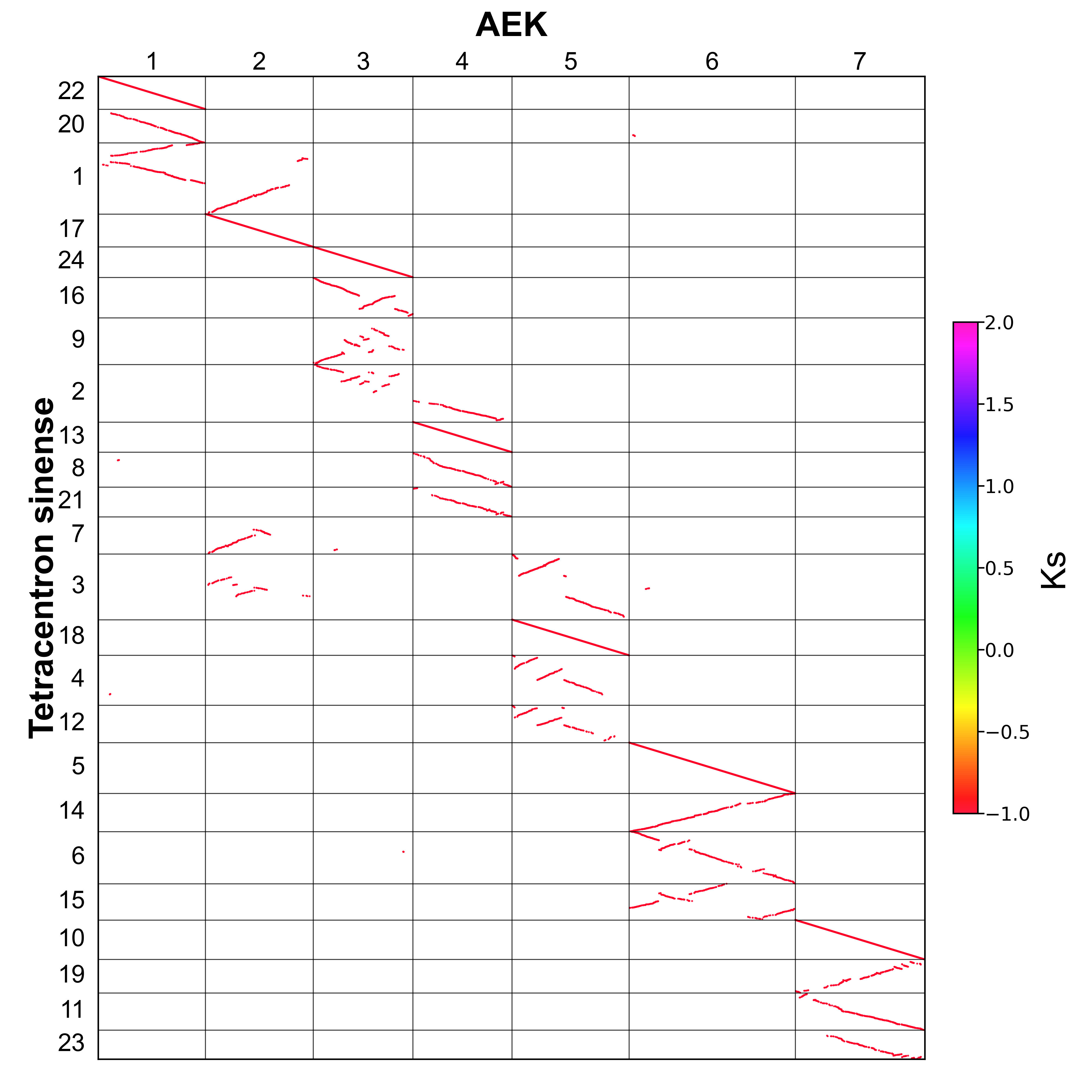
接下来又是一系列的计算:用 -icl(Improved collinearity)参数查找它们之间的共线性基因;用 -bi(BlockInfo)参数整合同源区块以便进行筛选;用 -c(Correspondence)参数滤除可能源自更古老进化事件的区块;最后用 -bk(BlockKs)参数验证剩余区块的合理性(图7)。5
图7 计算共线性块之间的 Ks 值,然后显示在原先的点图中。点迹变少主要来自提取共线性区块时零散共线性关系的丢失
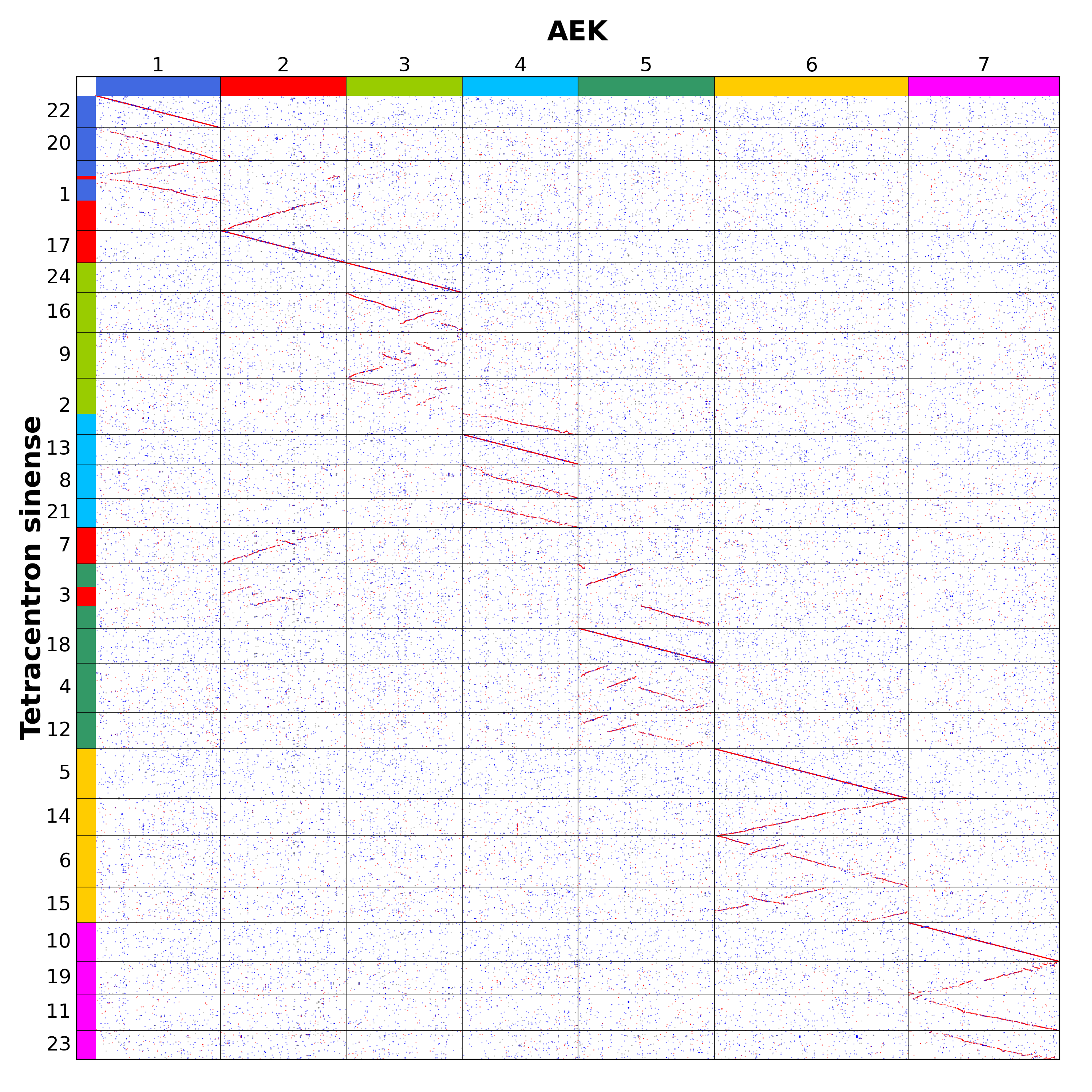
以使用 -km 参数生成的基因组与 AEK 映射关系作为补充文件,重新使用 -d 进行作图。此时可看到,与 AEK 同源的部分在左侧以不同颜色标出(图8)。
图8 使用 -km 结果重新绘制的点图
我们可以观察到,水青树的大部分基因与 AEK 存在共线性区块。同时,水青树的 Chr1 可通过 NCF 模式将 AEK1 插入 AEK2 后,再经 EEJ 模式与另一个 AEK1 融合形成,该过程使祖先染色体数量减少两条;其 Chr1 还可通过 EEJ 模式融合 AEK3 与 AEK4 形成,此过程使祖先染色体减少一条;其 Chr3 则可通过 NCF 模式将 AEK2 插入 AEK5 形成,该过程亦使祖先染色体减少一条。水青树从 AEK 经历两次全基因组倍增后理论上应具有 4×7=28 条染色体,当前通过 2+1+1=4 的减少过程实际保有24条染色体。未发现融合染色体重复现象,据此可推断水青树近期全基因组倍增前未发生染色体融合事件。水青树的核型演化过程与本研究提出的模型相符,这进一步验证了该模型的可靠性。
需指出的是,目前得到的该 AEK 结果尚且不能代表所有真双子叶植物的祖先核型,我们需要在其他物种中进一步验证。我们使用葡萄(欧亚葡萄,Vitis vinifera)来验证,依照先前流程,我们采用 WGDI 工具并设置参数 “-d, -icl, -bi,-c, -km, -d” 进行比对(图9)。
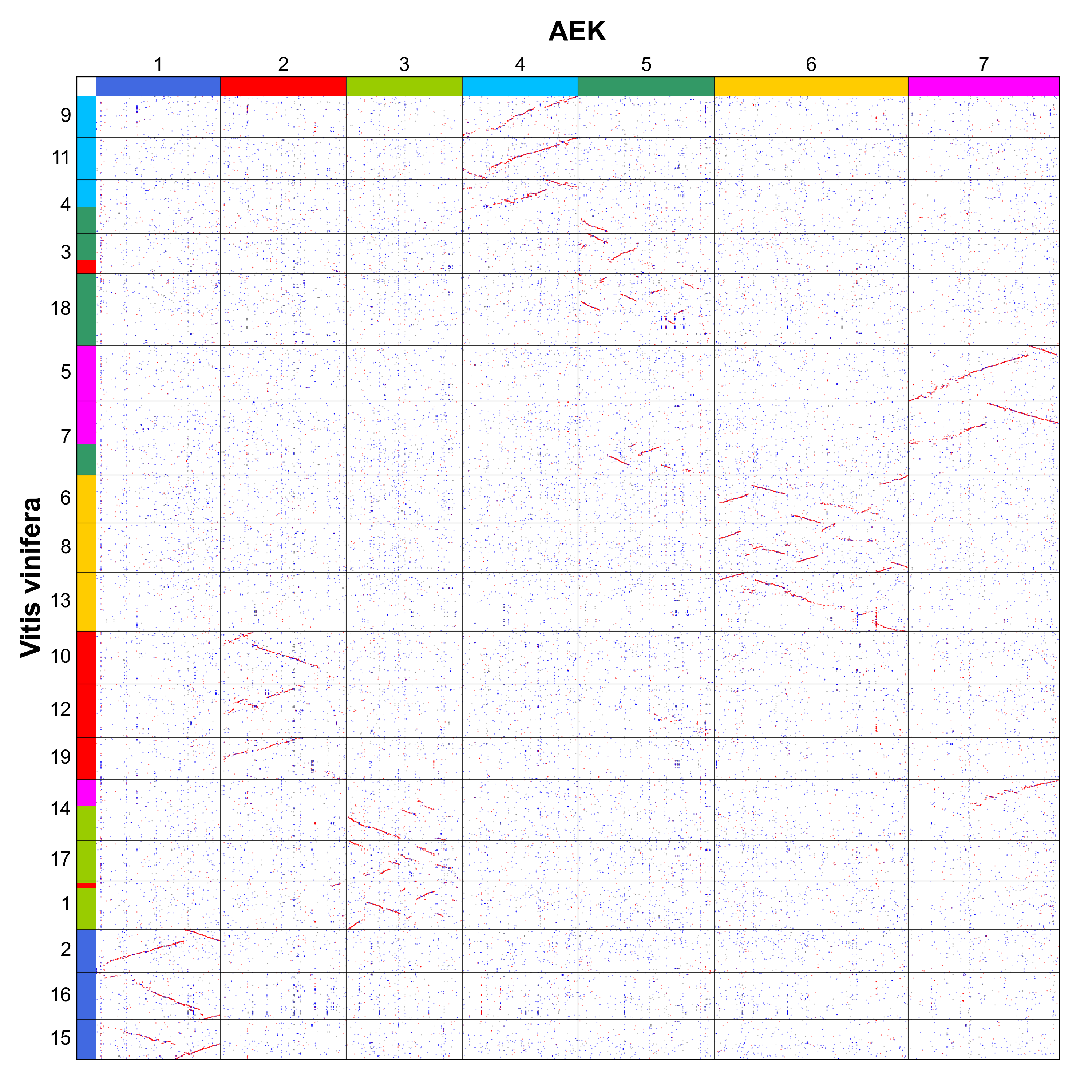
图9 欧亚葡萄与 AEK 的共线性关系点图
多倍性分析表明,葡萄存在全基因组三倍化事件,这意味着葡萄具有三份 AEK 拷贝。对于 AEK 的每个原染色体,均可在葡萄中发现完整的同源染色体,且不存在任何染色体构成原染色体的真子集。因此,这种包含 7 条原染色体的 AEK 的确可代表所有真双子叶植物的祖先核型。
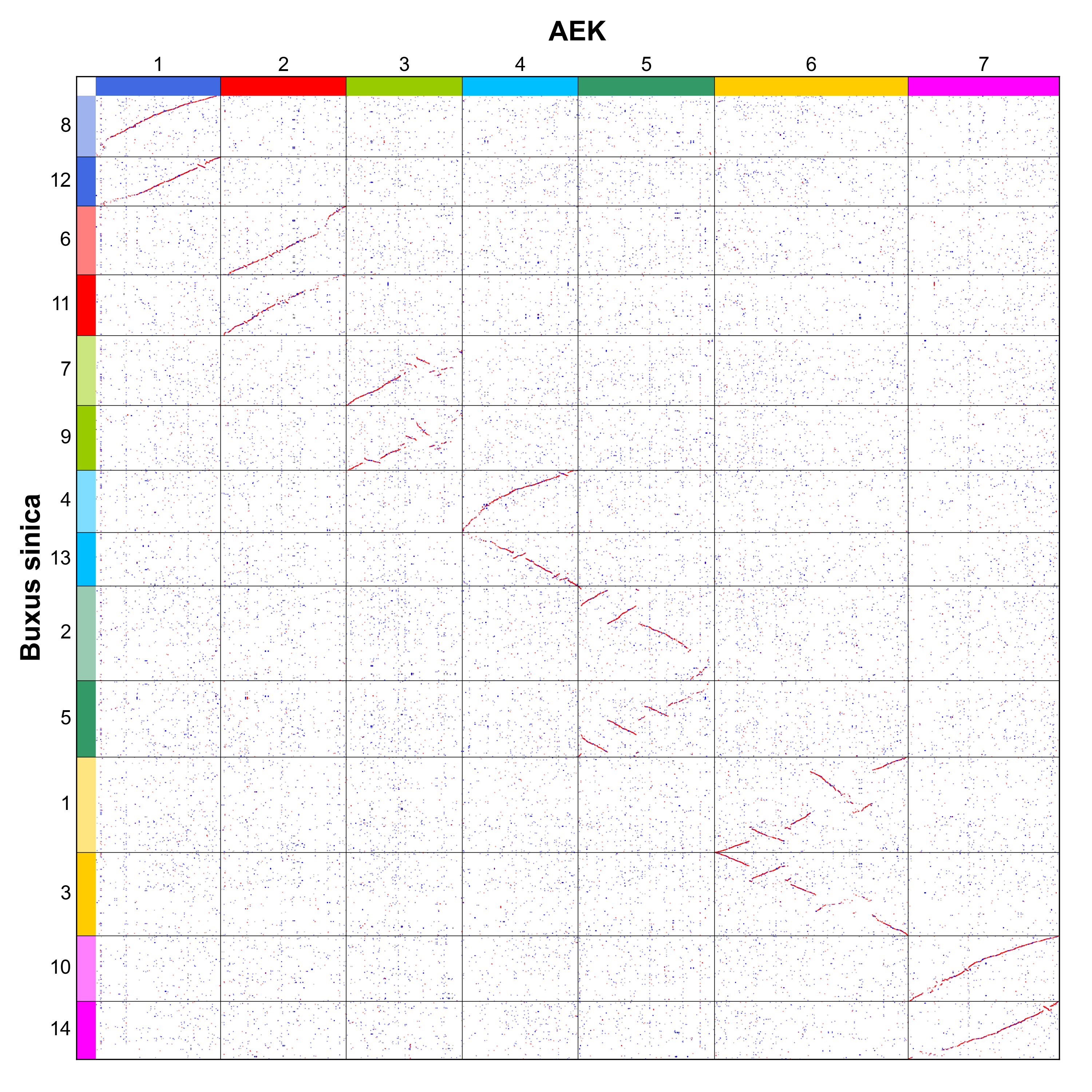
所选物种对于核型重建至关重要。部分物种具有稳定的染色体结构,且大多数染色体为原染色体。例如,中华黄杨(Buxus sinica)拥有14条染色体,并经历了独立的全基因组复制事件,其染色体在一次全基因组复制后直接从 AEK 保留而来。我们绘制了 AEK 与中华黄杨的同源点图,通过水青树推断的 AEK 与中华黄杨的结果一致,表明核型重建结果具有唯一性(图10)。
图10 中华黄杨与 AEK 的共线性关系点图
核型重建演示——以 ACEK为例
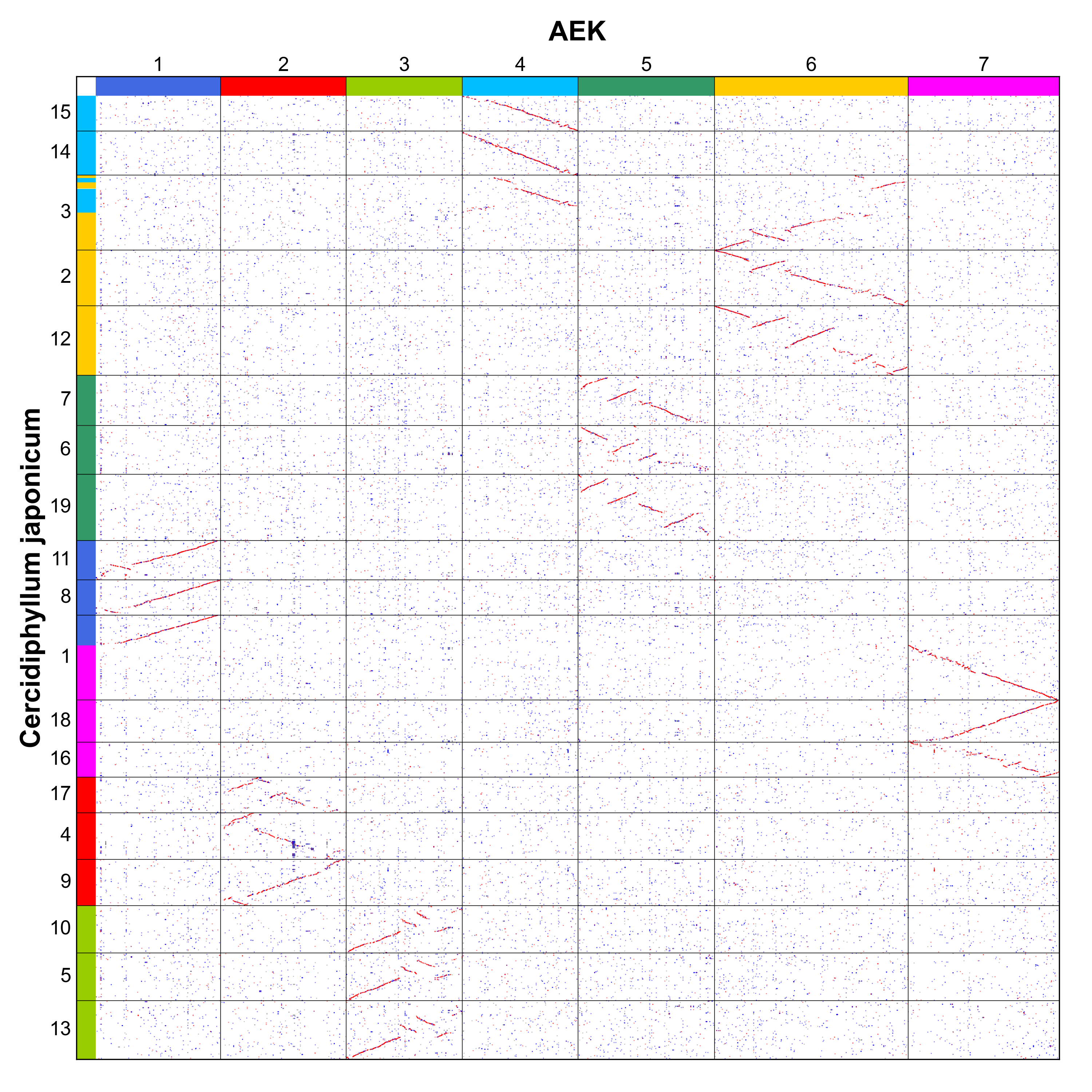
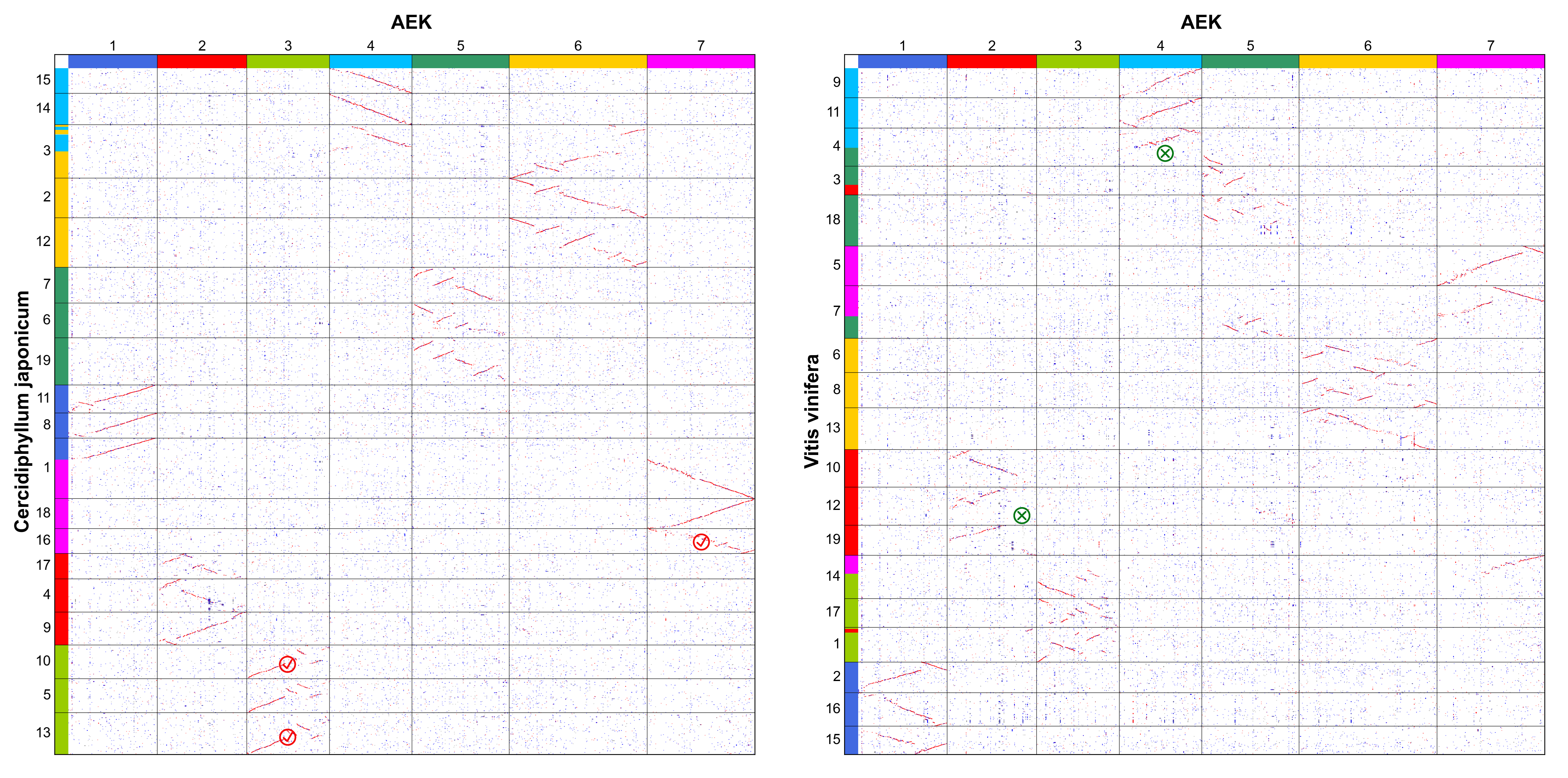
在经历全基因组三倍化后,ACEK6 应具有 3×7=21 条原染色体7。为探究这21条原染色体在进化过程中是否存在共有的染色体融合事件,我们在原有葡萄基因组参照基础上新增连香树(Cercidiphyllum japonicum)作为外群参照8。通过既有分析流程,可清晰追溯 AEK 原染色体在连香树基因组中的核型排布特征(图11)。
图11 连香树与 AEK 的共线性关系点图
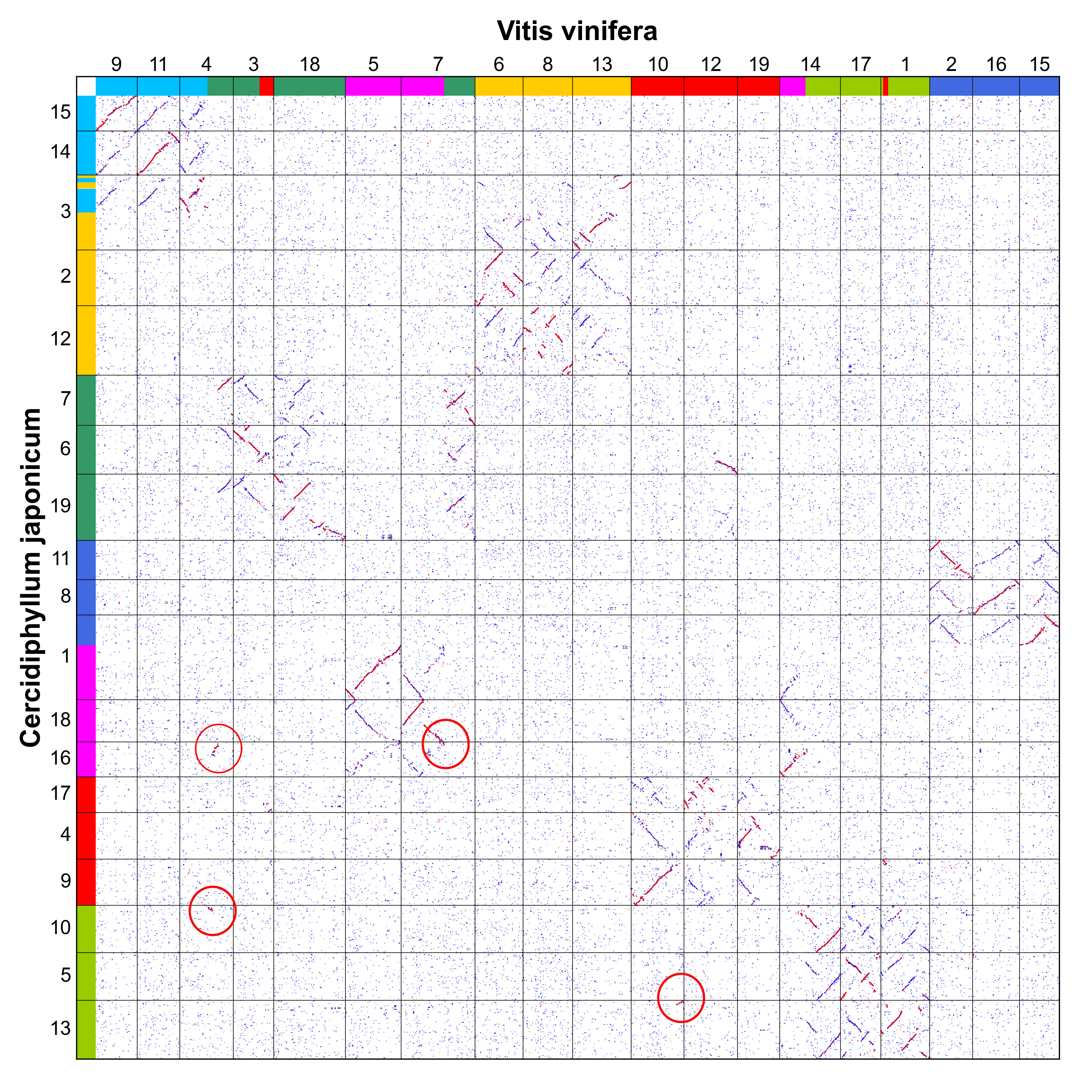
随后,我们使用 WGDI 工具的 -d 参数绘制了连香树(Cja)与葡萄(Vvi)间的同源点阵图。图中红点表示同源基因对为最佳匹配结果,由这些红点连接的共线性区块表明它们是物种分化后保留的旁系同源区块。源自相同原染色体的旁系同源区块采用统一颜色标识,但本点阵图中存在四处颜色不匹配现象(红色圆圈标注)(图12)。
图12 连香树与欧亚葡萄的共线性关系点图。红色圆圈中间出现的红色连线属于异常的狭窄共线性区段
这是因为某些结构变异的小片段与 AEK 之间缺乏高质量的共线性片段。针对这些情况,我们选择未发生结构变异的整条染色体作为 ACEK 的原染色体,且优先选择来自连香树的染色体(因为连香树的核型显然更接近 ACEK)。
例如:
- 对于 AEK1(蓝),由于 Cja1 的蓝色条带对应完整染色体 Vvi15,我们选择 Cja8、Cja11 和 Vvi15;
- 对于 AEK2(红),由于葡萄红色条带的分散分布,我们选择 Cja17、Cja4 和 Cja9;
- 对于 AEK3(草绿),由于葡萄草绿色条带的分散分布,我们选择 Cja10、Cja5 和Cja13;
- 对于 AEK4(天蓝),由于连香树天蓝色条带的不连续分布,我们选择 Cja15、Cja14 及 Vvi4 的天蓝色条带;
- 对于 AEK5(深绿),由于葡萄深绿色条带的分散分布,我们选择 Cja7、Cja6 和 Cja19;
- 对于 AEK6(橙黄),由于连香树橙黄色条带的不连续分布,我们选择 Vvi6、Vvi8 和 Vvi13;
- 对于 AEK7(紫),由于 Cja1 的紫色条带对应完整染色体 Vvi5,我们选择 Vvi5、Cja18 和 Cja16(参考图12和图 13)。
该过程中未发现共享的融合事件,故分化前的 ACEK 数量为 21。
图13 比较连香树、欧亚葡萄与 AEK 的共线性关系点图,以选择合适的染色体重组 ACEK。图中的红色对号与绿色叉号表示后代的这条染色体是否与 AEK 的完全匹配
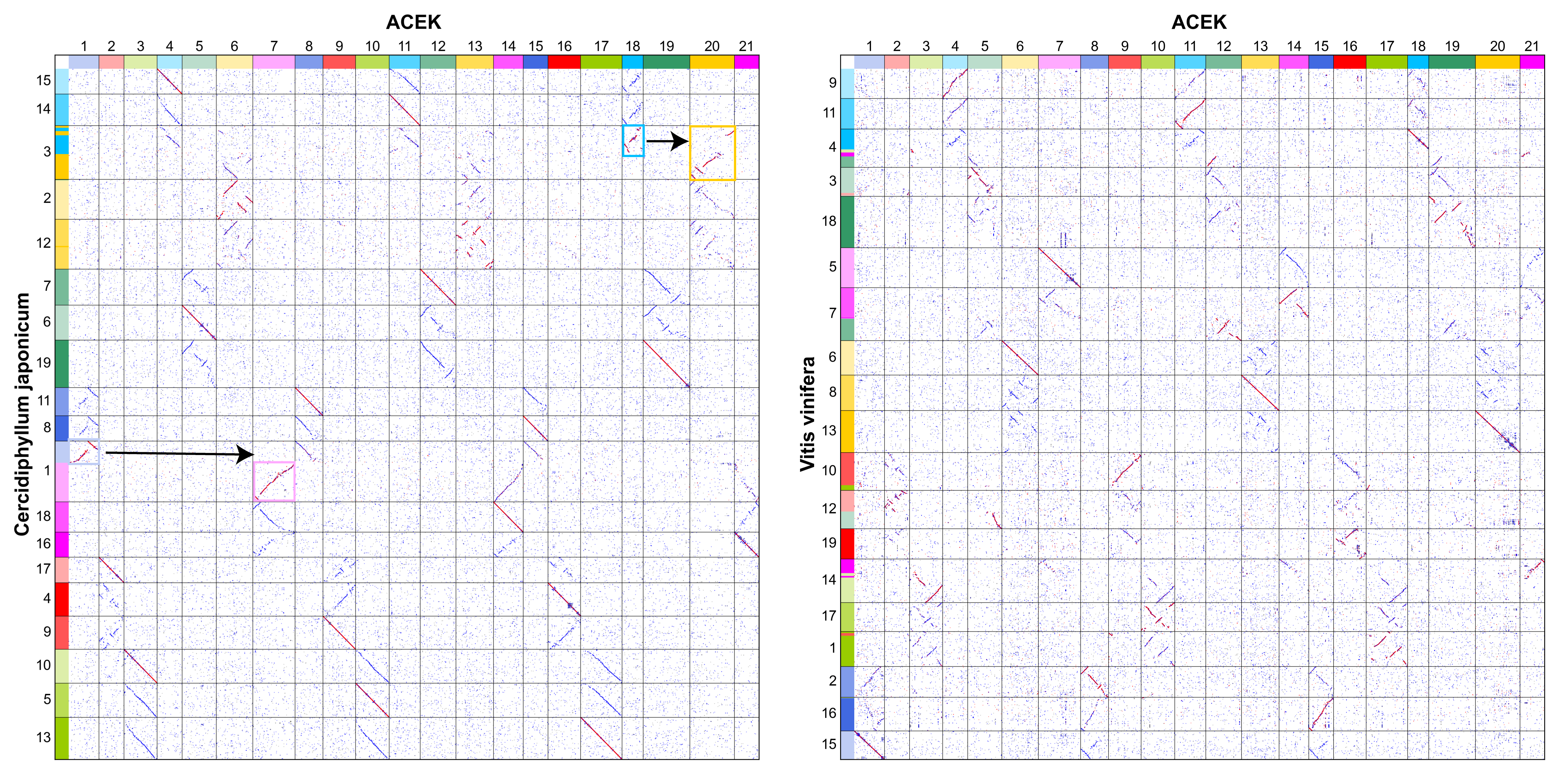
最终,我们将自己的 ACEK 映射至连香树和葡萄基因组。就连香树而言,自 ACEK 分化后经历两次融合事件:通过 NCF 模式将 ACEK18 插入 ACEK20(右上箭头),以及通过 EEJ 模式融合 ACEK1 与 ACEK7(左下箭头)(图14)。
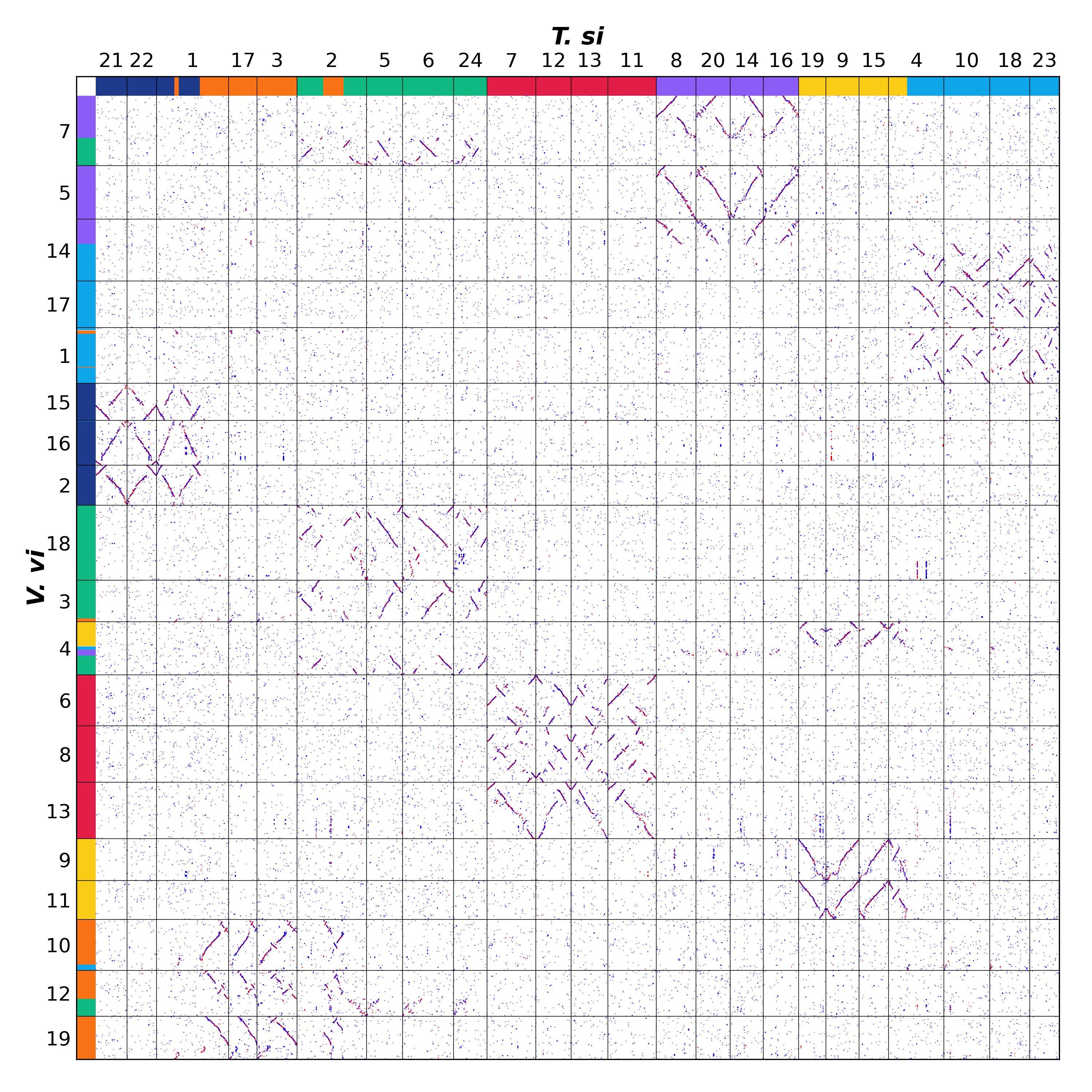
图14 连香树、欧亚葡萄与自构建 ACEK 的共线性点图
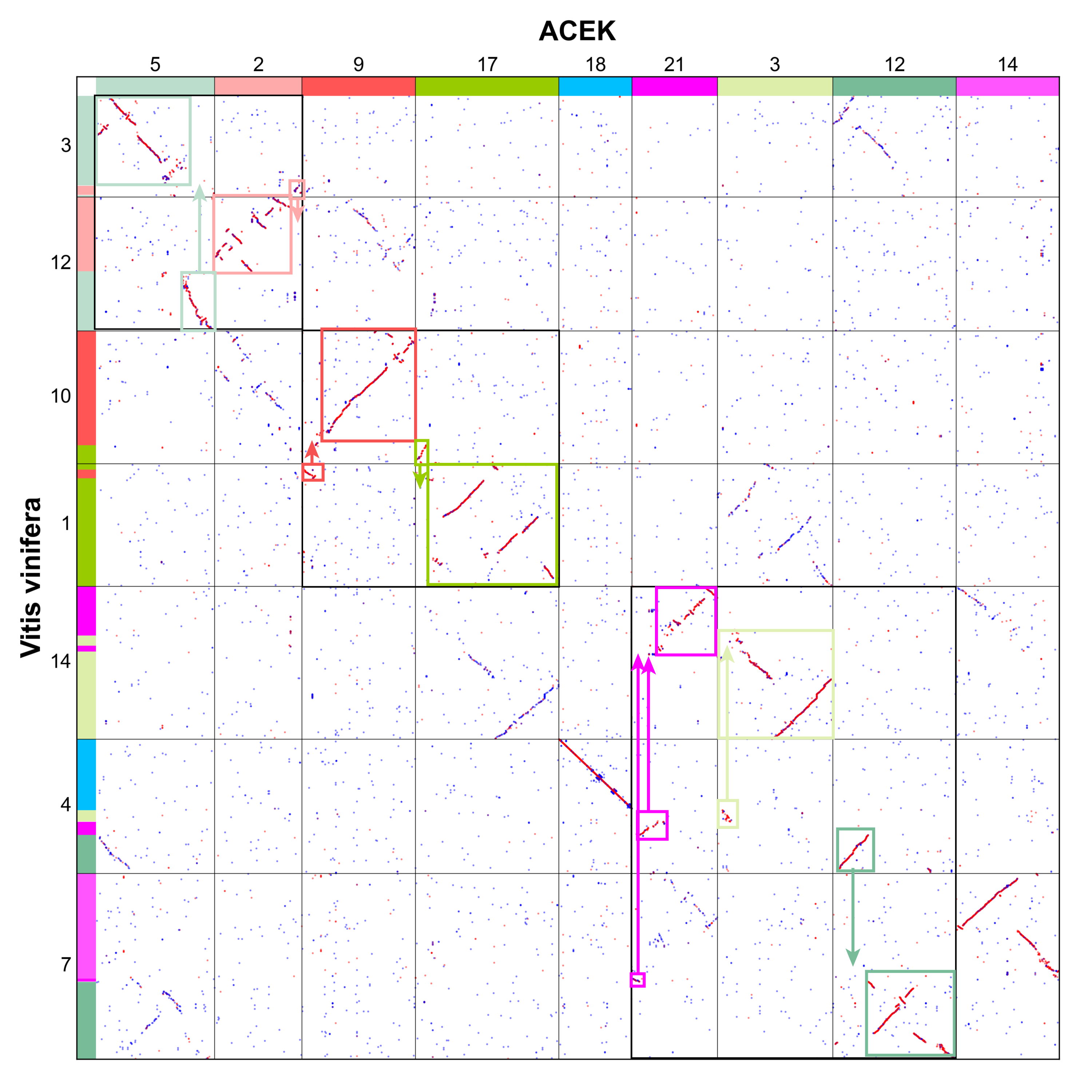
对葡萄而言,为便于追溯其祖先染色体变异,我们对涉及重排的染色体重新绘制了点阵图(图15)。Vvi3+12 完全等同于 ACEK5+2,该现象与我们建立的 RTA 模型相符。若假设两条染色体先发生断裂再融合,断裂产生的四条染色体更易与数量更多的其他染色体结合。因此在此采用 RTA 模型更具合理性。通过 RTA 模型,ACEK9 与 ACEK17 融合形成两条染色体(Vvi10 和 Vvi1);ACEK21 与 ACEK3 融合为一条染色体(Vvi14)及一段微小染色体(Vvi4 中段);该微小染色体又与 ACEK12 进一步融合形成两条染色体(Vvi4 部分区段与 Vvi7 部分区段)。这两条染色体后期分别与 ACEK18、ACEK14 融合为Vvi4 和 Vvi7。
葡萄的染色体演化过程较为复杂,特别是 ACEK21、ACEK3 与 ACEK12之间(对应葡萄第 14、4 和 7 号染色体)存在的变异易引发可靠性质疑(图15)。值得庆幸的是,葡萄属另一亚属的研究数据为我们提供了有力佐证。
图15 对葡萄基因组中发生重排的染色体重做点图。部分染色体(特别是葡萄的 14、4 和 7 号染色体)与 ACEK 的关系复杂,难以阐述,也难以证明
葡萄属两个亚属的动态演化历史
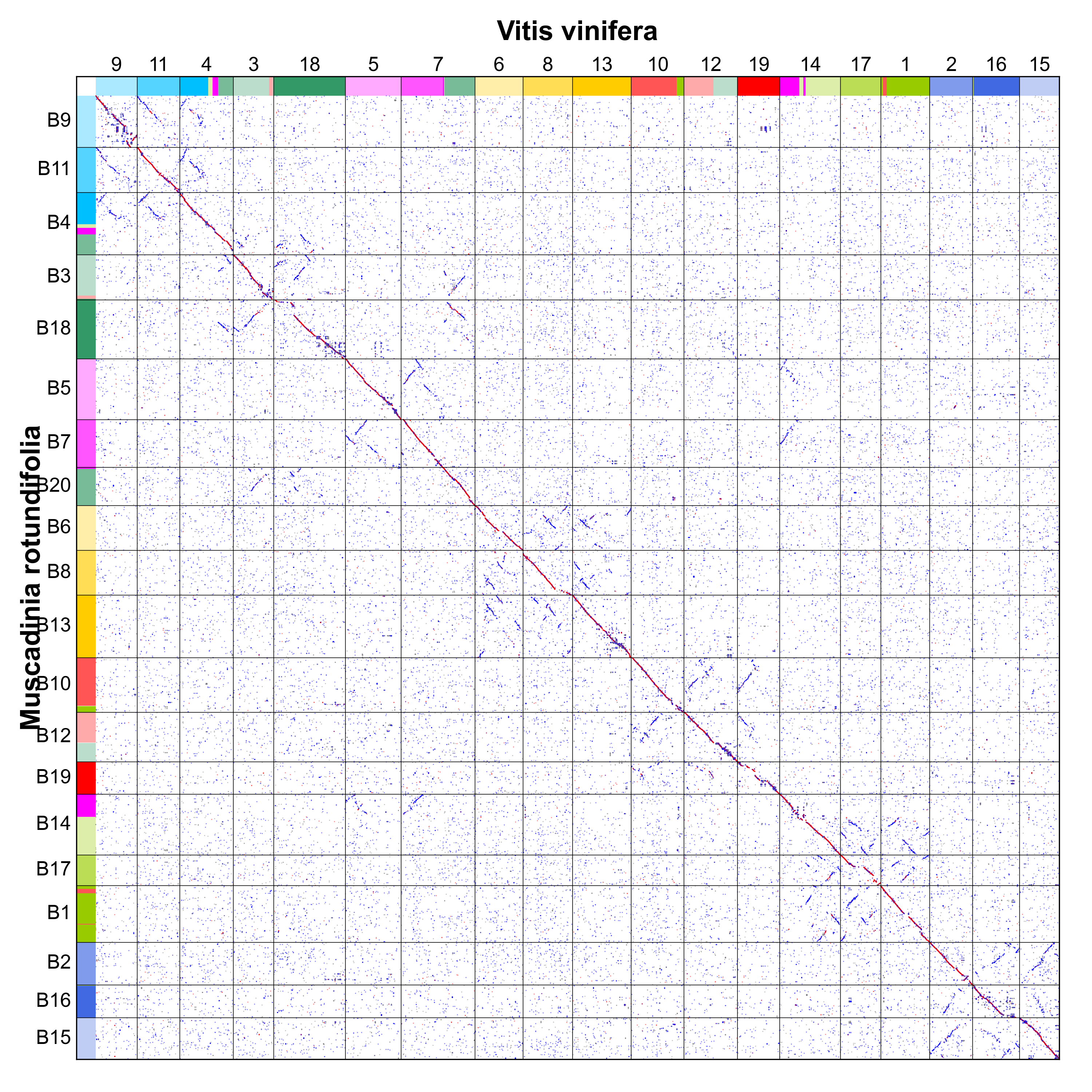
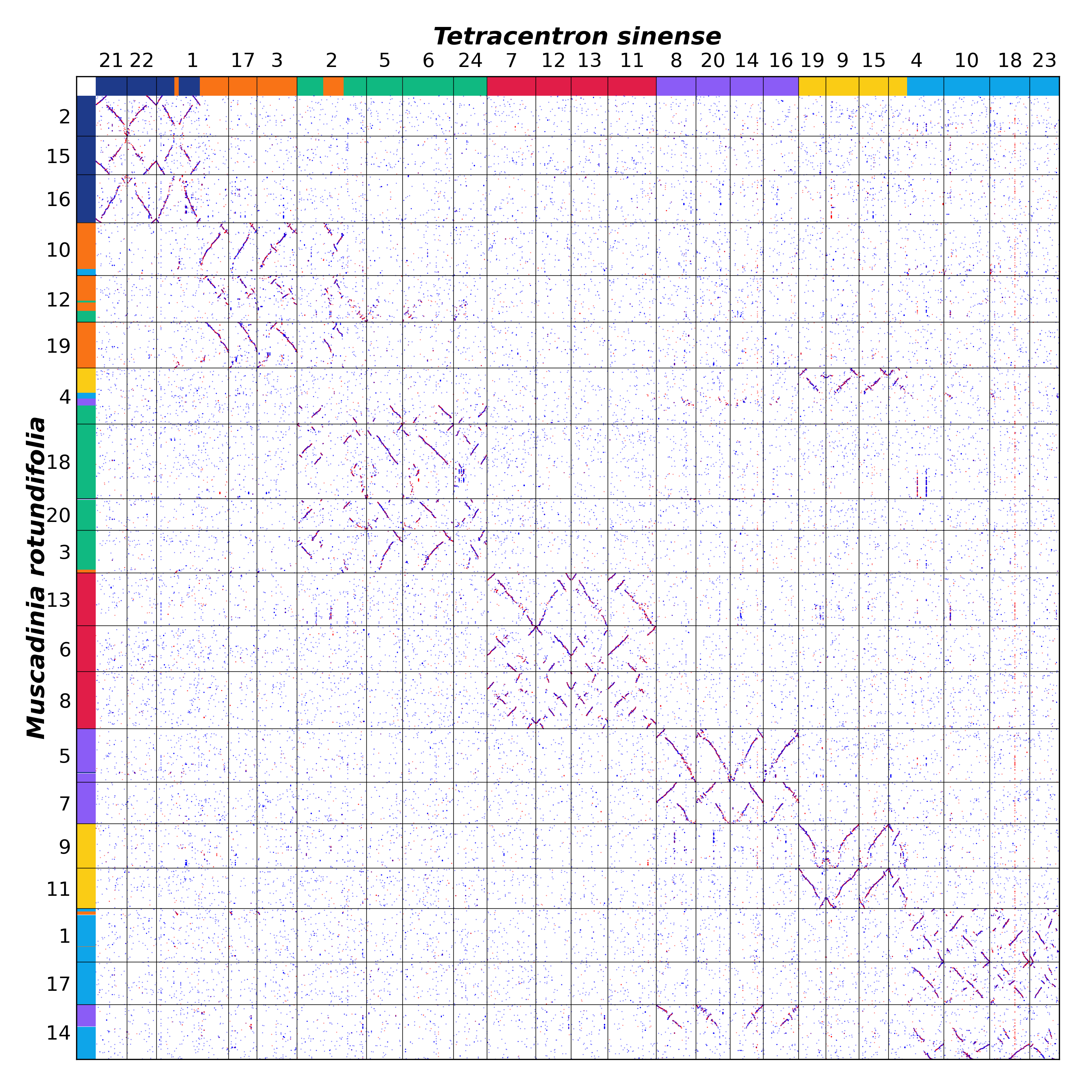
葡萄属分为麝香葡萄亚属(Muscadinia Planch,2n=40)和真葡萄亚属(Euvitis Planch,2n=38)。圆叶葡萄(Muscadinia rotundifolia)属于麝香葡萄亚属,而欧亚葡萄(V. vinifera)属于真葡萄亚属。我们以相同方式获得了圆叶葡萄的祖先十字花科核型定位(图16)。
图16 圆叶葡萄与 ACEK 的共线性点图
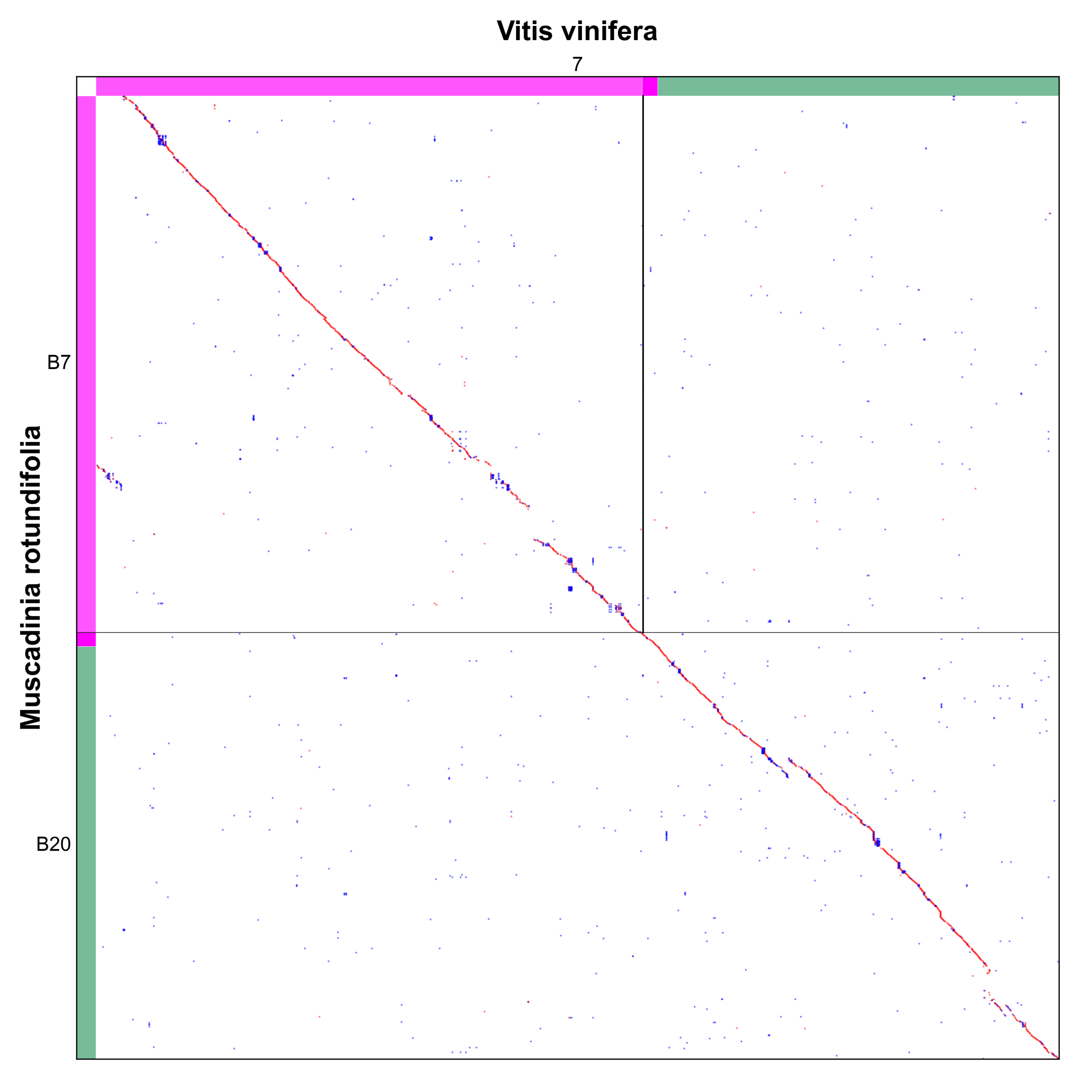
比较欧亚葡萄与圆叶葡萄时发现(图17和图18),大多数祖先染色体融合事件在葡萄属中具有共享性。唯一差异体现在 MroB7、MroB20 与 Vvi7 之间。
假设 MroB7 和 MroB20 由 Vvi7 分裂形成。但 Vvi7 来自 ACEK12+14,要让它再分裂回 MroB7(=ACEK14),显然不太可能。因此,将 MroB7 与 MroB20 融合为 Vvi7 更为合理。
同时,MroB14 未发生 ACEK 框架下的倒位现象。这些证据均表明,圆叶葡萄可能更接近葡萄属的祖先核型。
图17 圆叶葡萄与欧亚葡萄的共线性点图,包含两个物种基因组到 ACEK 的映射关系
图18 圆叶葡萄 B7 号(MroB7)、B20 号(MroB20)染色体与欧亚葡萄 7 号(Vvi7)染色体的微观共线性点图
总而言之,葡萄属 ACK 从 ACEK 开始的动态演化过程如下(图19):
图19 葡萄属核型从 ACEK 开始的动态演化过程。两种葡萄的 12 号和 3 号染色体都是 ACEK 5 号和 2 号染色体发生 RTA 的结果(a),10 号和 1 号染色体来自 ACEK 9 号和 17 号,也经历了类似过程,但 1 号染色体的交换断点处又发生了一次倒位(b)。比对两种葡萄基因组到 ACEK 的映射,无论是欧亚葡萄在原有的交换断点处发生倒位(MroB14->Vvi14),还是融合了两个非同源染色体(MroB7+MroB20->Vvi7),都证明圆叶葡萄比欧亚葡萄更加原始(c)。由此,葡萄属 14、4 和 7 号染色体形成过程的假说得到证实
该核型演化模型能够清晰阐明这四个物种的核型演化过程,特别是葡萄属两个亚属的动态演化历史。这使我们有充分理由相信,该模型很可能同样适用于所有被子植物。
实践与结果复现
这一部分并非来自原文作者,而是我采用自己搜索到的数据进行的结果重现。
重建 AEK
使用的是水青树的参考基因组GCA_015143295.1](https://www.ncbi.nlm.nih.gov/datasets/genome/GCA_015143295.1/)。自共线性结果看上去还不错,甚至优于作者的(图20)。
图20 使用水青树参考基因组建立的自共线性点图,结果与预期一致
ACK 的颜色标识是:
| 颜色 | 十六进制值 | ACK 代号 |
|---|---|---|
| 深蓝 | #1E3A8A | ACK1 |
| 橙 | #F97316 | ACK2 |
| 青绿 | #10B981 | ACK3 |
| 洋红 | #E11D48 | ACK4 |
| 紫罗兰 | #8B5CF6 | ACK5 |
| 金黄 | #FACC15 | ACK6 |
| 天蓝 | #0EA5E9 | ACK7 |
重建 ACEK
由于找不到连香树的基因组,我选择只使用葡萄(V. vinifera)的参考基因组GCF_030704535.1。先对葡萄和水青树做 blast、做共线性,再将原本水青树到 AEK 的映射延伸到葡萄上。可以看到,结果出现了细微偏差:葡萄的 Chr1 和 Chr 16 各自多出了一个条带;Chr 14 融合断点处的倒位也不见了(图21)。
图21 葡萄与水青树的共线性点图,彩条表示各自所属的 AEK 原染色体
检查一下葡萄基因组的 ancestor map(节选):
1
2
3
4
5
6
7
1 84 187 #F97316 1
1 188 1145 #0EA5E9 1
1 1146 1160 #F97316 1
1 1161 1620 #0EA5E9 1
16 1 23 #0EA5E9 1
16 24 1294 #1E3A8A 1
1 1 83 #0EA5E9 1
插入了大约 20 个同源序列对,数量占 2% 左右。——只使用葡萄的缺点出现了。
不行。还得拉圆叶葡萄出来。圆叶葡萄比葡萄原始一些,结果估计也更加清晰一点。
遗憾的是,NCBI 数据库的圆叶葡萄参考基因组没有注释。于是我们选择了 2023 年 Matthew Huff 等人发表的基因组 GCA_030254885.1,该基因组基于圆叶葡萄 “Carlos” 品种,质量也算不错,就是 Contig N50 差了些(完整性 97.7%,Contig N50 11.3 Mb,Scaffold N50 20.8 Mb,测序深度 281x)9。
将上面那一套给它用上。
图22 圆叶葡萄与水青树的共线性点图
5 号和12 号染色体上仍然有噪声区段。不过 20 号那里的紫色没有和其他紫色染色体结合,而葡萄 7 号染色体的紫色片段来自两个原染色体且难以区分。——确实有奇效!
那么接下来,以圆叶葡萄为锚,参考葡萄的结果重新做一下 map 吧。
- 深蓝色
#1E3A8A(2、15、16)、红色#E11D48(13、6、8)和黄色#FACC15(4、9、11)照抄; - 橙色
#F97316取 19 号、10 号橙色 + 1 号橙色、12 号橙色 + 3 号橙色; - 绿色
#10B981取 18 号、20 号绿色 + 4 号绿色、3 号绿色 + 12 号绿色; - 紫色
#8B5CF6取 5 号、7 号、14 号紫色 + 4 号紫色 + 20 号紫色; - 蓝色
#0EA5E9取 17 号、1 号蓝色 + 10 号蓝色、14 号蓝色 + 4 号蓝色。
顺序与结构都不重要,只要准确划分到 ACEK 各组的不同原染色体即可。取色时,第一个取原色,第二个取原色 80%,第三个取 50%。
ACEK 的颜色标识是:
| 颜色 | 十六进制值 | ACEK 代号 |
|---|---|---|
| 深蓝 | #1E3A8A #4B61A1 #8E9CC4 | ACEK1-3 |
| 橙 | #F97316 #FA8F45 #FCB98A | ACEK4-6 |
| 青绿 | #10B981 #40C79A #88DCC0 | ACEK7-9 |
| 洋红 | #E11D48 #E74A6D #F08EA4 | ACEK10-12 |
| 紫罗兰 | #8B5CF6 #A27DF8 #C5AEFA | ACEK13-15 |
| 金黄 | #FACC15 #FBD644 #FCE68A | ACEK16-18 |
| 天蓝 | #0EA5E9 #3EB7ED #86D2F4 | ACEK19-21 |
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
1 1 574 #1E3A8A 1
1 575 715 #F97316 1
1 716 1405 #1E3A8A 1
1 1406 2347 #F97316 1
10 1 1498 #0EA5E9 1
11 1 1578 #E11D48 1
12 1 1153 #E11D48 1
13 1 1193 #E11D48 1
14 1 1086 #8B5CF6 1
15 1 963 #FACC15 1
16 1 1149 #8B5CF6 1
17 1 926 #F97316 1
18 1 1303 #0EA5E9 1
19 1 887 #FACC15 1
2 1 850 #10B981 1
2 851 1515 #F97316 1
2 1516 2260 #10B981 1
20 1 1110 #8B5CF6 1
21 1 1020 #1E3A8A 1
22 1 964 #1E3A8A 1
23 1 960 #0EA5E9 1
24 1 1097 #10B981 1
3 1 1301 #F97316 1
4 1 597 #FACC15 1
4 598 1792 #0EA5E9 1
5 1 1179 #10B981 1
6 1 1653 #10B981 1
7 1 1581 #E11D48 1
8 1 1292 #8B5CF6 1
9 1 1081 #FACC15 1
将这些信息写到 map 上,再走一下 -km -ak 和 -akr,就成功了。
具体方法如下:
- 将这个 map 映射到圆叶葡萄基因组(
-km),得到圆叶葡萄到祖先的完整 map; - 使用
-km转换为祖先染色体核型; - 再使用
-akr补充葡萄的基因信息。
不过需要注意,如果直接使用只有 6 列的 gff,它就会报错:
Traceback (most recent call last): File "/home/hyli360/miniconda3/envs/py312/lib/python3.12/site-packages/pandas/core/indexes/range.py", line 413, in get_loc return self._range.index(new_key) ^^^^^^^^^^^^^^^^^^^^^^^^^^ ValueError: 6 is not in range最简单的解决方法是,在最后一列加点东西:
只需第一行加上即可。
原理:
出错的这一步位于
原本是用来检查
.gff(df)的第 6 列是否在.faa(pep)的 keys 里。原因在于-ak和-akr接受的是 7 列(带旧名字的)GFF,而不是最原始的 6 列 GFF。该解决方法不会对后续结果造成影响。
最后再做一下点图,检验成果。
图23 圆叶葡萄基因组与自组建 ACEK 的共线性点图
图24 葡萄基因组与自组建 ACEK 的共线性点图(左侧代表 AEK 核型,而非 ACEK 核型)
成功复现原文结果。
也叫“原染色体”(Protochromosome),与前文 Ancestor Chromosome 基本同义。原文中“祖先染色体”与“原染色体”两词混用,故直接翻译不做替换。 ↩︎
《纪念孟德尔、麦克林托克与达林顿:论染色体末端融合与嵌套融合》(Celebrating Mendel, McClintock, and Darlington: On end-to-end chromosome fusions and nested chromosome fusions),载于 Plant Cell 期刊。https://doi.org/10.1093/plcell/koac116 ↩︎
即 Ancestral eudicots karyotypes,双子叶植物祖先染色体核型。 ↩︎
较原始的双子叶植物,也是真双子叶植物四大早期分化支系之一。 ↩︎
即 Ancestral core eudicots karyotypes,核心真双子叶植物祖先染色体核型。 ↩︎
这一变化主要来自距今1.2亿年前的“γ 三倍化事件”,简单来说,ACEK 是 AEK 三倍化后重排产生的结果。几乎所有核心真双子叶植物都带有这个痕迹,其中葡萄的三倍化核型较为稳定,更接近 ACEK,是研究核心真双子叶植物核型演化的基本参考。 ↩︎
连香木在系统发育上接近核心真双子叶基部,而葡萄属于十字花科,位于核心真双子叶植物类群的更深处。 ↩︎